Python 中的乳腺癌分类项目
了解 Python 中乳腺癌分类项目中使用的术语
什么是深度学习?
深度学习是一种密集的机器学习方法,其灵感来自人脑及其生物神经网络的工作原理。深度神经网络、递归神经网络、卷积神经网络和深度信念网络等架构由多层组成,供数据在最终生成输出之前通过。深度学习有助于改进人工智能,使其许多应用成为可能;它应用于计算机视觉、语音识别、自然语言处理、音频识别和药物设计等许多此类领域。
什么是Keras?
Keras 是一个用 Python 编写的开源神经网络库。它是一个高级 API,可以在 TensorFlow、CNTK 和 Theano 之上运行。Keras 旨在实现快速实验和原型设计,同时在 CPU 和 GPU 上无缝运行。它是用户友好的、模块化的和可扩展的。
乳腺癌分类 – 目标
在 IDC 数据集上构建乳腺癌分类器,该分类器可以准确地将组织学图像分类为良性或恶性。
乳腺癌分类 – 关于 Python 项目
在这个 python 项目中,我们将构建一个分类器来训练 80% 的乳腺癌组织学图像数据集。其中,我们将保留 10% 的数据进行验证。使用 Keras,我们将定义一个 CNN(卷积神经网络),将其命名为 CancerNet,并在我们的图像上对其进行训练。然后,我们将推导出一个混淆矩阵来分析模型的性能。
IDC 是浸润性导管癌;在乳腺导管中发展并侵入乳腺导管外的纤维或脂肪乳腺组织的癌症;它是最常见的乳腺癌形式,占所有乳腺癌诊断的 80%。组织学是对组织微观结构的研究。
数据集
我们将使用 Kaggle 的 IDC_regular 数据集(乳腺癌组织学图像数据集)。该数据集包含 2,77,524 个大小为 50×50 的斑块,这些斑块是从 162 张以 40 倍扫描的乳腺癌标本的整个载玻片图像中提取的。其中,IDC 检测结果为阴性 1,98,738 人,78,786 人检测呈阳性。该数据集在公共领域可用,您可以在此处下载。为此,您至少需要 3.02GB 的磁盘空间。
此数据集中的文件名如下所示:
8863_idx5_x451_y1451_class0
这里,8863_idx5 是患者 ID,451 和 1451 是作物的 x 和 y 坐标,0 是类标签(0 表示没有 IDC)。
先决条件
您需要安装一些 python 包才能运行此高级 python 项目。你可以用 pip- 来做到这一点
pip install numpy opencv-python pillow tensorflow keras imutils scikit-learn matplotlib
Python 高级项目的步骤 – 乳腺癌分类
1. 下载此 zip。在您喜欢的位置解压缩,到达那里。
截图:
2. 现在,在内部乳腺癌分类目录中,创建目录数据集 - 在此目录中,创建原始目录:
mkdir 数据集mkdir 数据集\原始
3. 下载数据集。
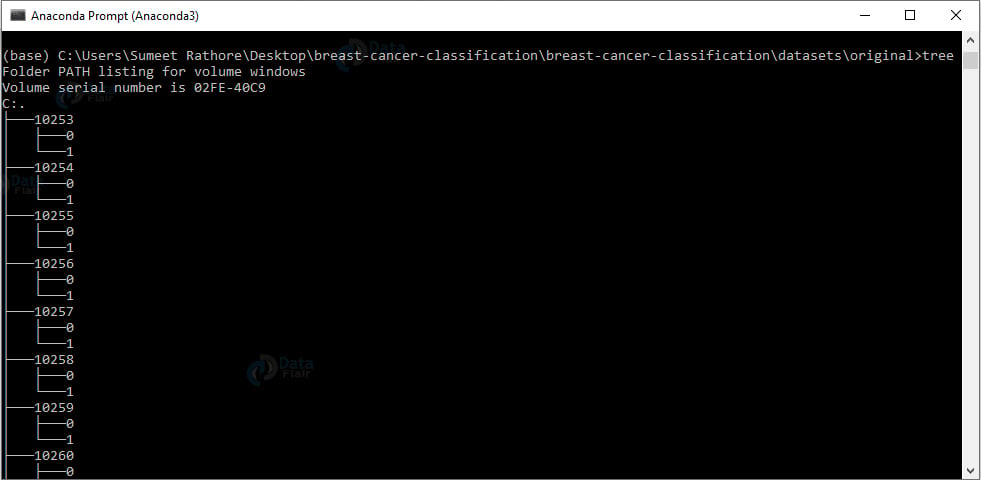
4. 解压原始目录下的数据集。要观察此目录的结构,我们将使用 tree 命令:
输出截图:
我们为每个患者 ID 提供了一个目录。在每个这样的目录中,我们都有 0 和 1 目录,用于具有良性和恶性内容的图像。
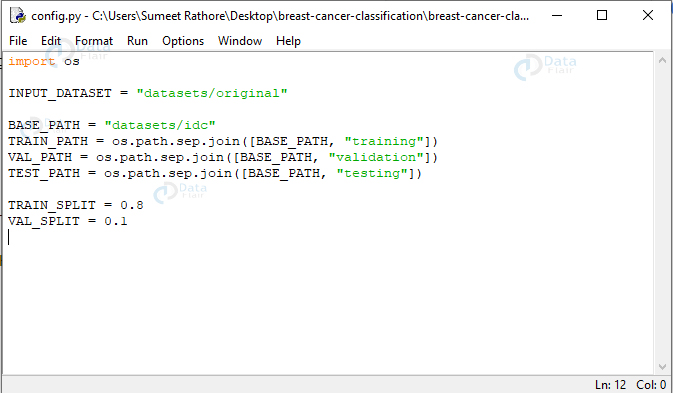
config.py:
这包含了构建数据集和训练模型所需的一些配置。您可以在 cancernet 目录中找到它。
INPUT_DATASET = “数据集/原始”BASE_PATH = “数据集/IDC”TRAIN_PATH = 操作系统。路径。9月join([BASE_PATH, “训练”])VAL_PATH = 操作系统。路径。9月join([BASE_PATH, “验证”])TEST_PATH = 操作系统。路径。9月join([BASE_PATH, “测试”])TRAIN_SPLIT = 0.8VAL_SPLIT = 0.1
截图:
在这里,我们声明输入数据集的路径 (datasets/original)、新目录的路径 (datasets/idc) 以及使用基本路径的训练、验证和测试目录的路径。我们还声明整个数据集的 80% 将用于训练,其中 10% 将用于验证。
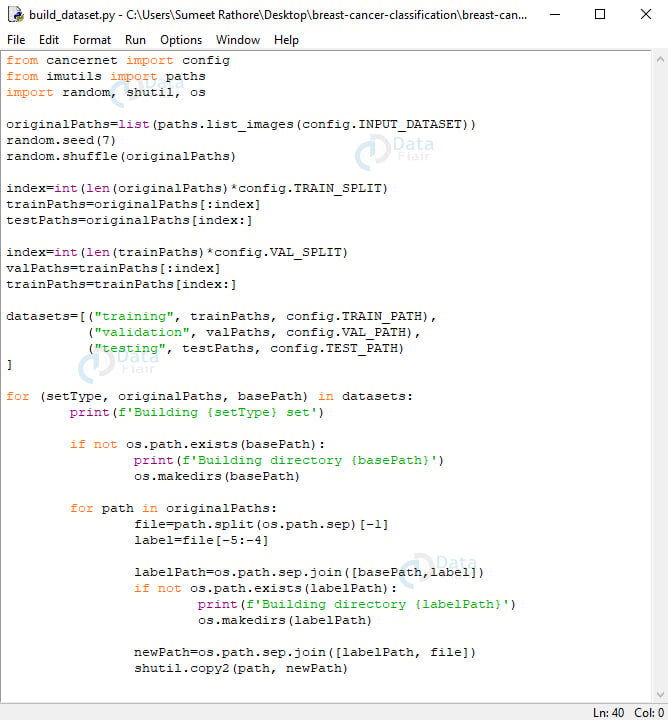
build_dataset.py:
这会将我们的数据集按上述比例分为训练集、验证集和测试集——80% 用于训练(其中 10% 用于验证),20% 用于测试。使用 Keras 的 ImageDataGenerator,我们将提取批量图像,以避免一次在内存中为整个数据集腾出空间。
从 cancernet 导入配置从 imutils 导入路径导入随机、shutil、操作系统originalPaths=list(paths.list_images(config.INPUT_DATASET))随机。种子(7)随机。shuffle(原始路径)index=int(len(originalPaths)*配置。TRAIN_SPLIT)trainPaths=originalPaths[:index]testPaths=originalPaths[索引:]index=int(len(trainPaths)*config。VAL_SPLIT)valPaths=trainPaths[:index]trainPaths=trainPaths[index:]datasets=[(“training”, trainPaths, config.TRAIN_PATH),(“validation”, valPaths, config.VAL_PATH),(“testing”、testPaths、配置。TEST_PATH)]for (setType, originalPaths, basePath) 在数据集中:print(f'构建 {setType} set')如果不是 OS。路径。存在(basePath):print(f'构建目录 {base_path}')操作系统。makedirs(basePath))对于 originalPaths 中的 path:file=路径。split(os.路径。9月)[-1]标签=文件[-5:-4]labelPath=os。路径。9月join([basePath,label])如果不是 OS。路径。存在(labelPath):广告print(f'构建目录 {labelPath}')
操作系统。makedirs(labelPath))newPath=os。路径。9月join([labelPath, 文件])舒蒂尔。copy2(输入路径, newPath)
截图:
在这里,我们将从 config、imutils、random、shutil 和 os 导入。我们将构建图像的原始路径列表,然后对列表进行随机排序。然后,我们通过将此列表的长度乘以 0.8 来计算索引,以便我们可以对该列表进行切片以获得训练和测试数据集的子列表。接下来,我们进一步计算一个指数,将 10% 的列表用于训练数据集进行验证,并将其余部分保留用于训练本身。
现在,数据集是一个包含元组的列表,用于提供有关训练集、验证集和测试集的信息。它们保存每个路径和基本路径。对于此列表中的每个 setType、路径和基本路径,我们将打印“构建测试集”。如果基本路径不存在,我们将创建目录。对于 originalPaths 中的每个路径,我们将提取文件名和类标签。我们将构建标签目录(0 或 1)的路径 - 如果它还不存在,我们将显式创建此目录。现在,我们将构建生成图像的路径,并将图像复制到它所属的位置。
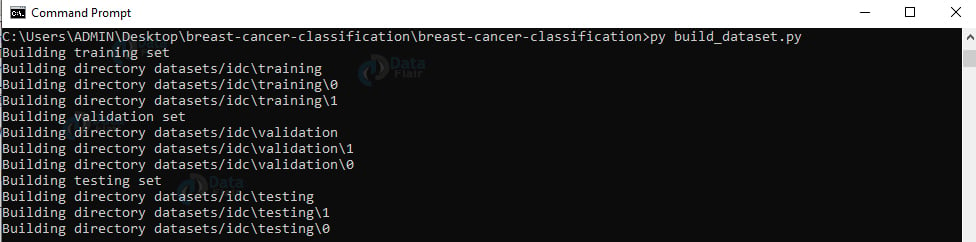
5. 运行脚本build_dataset.py:
py build_dataset。py
输出截图:
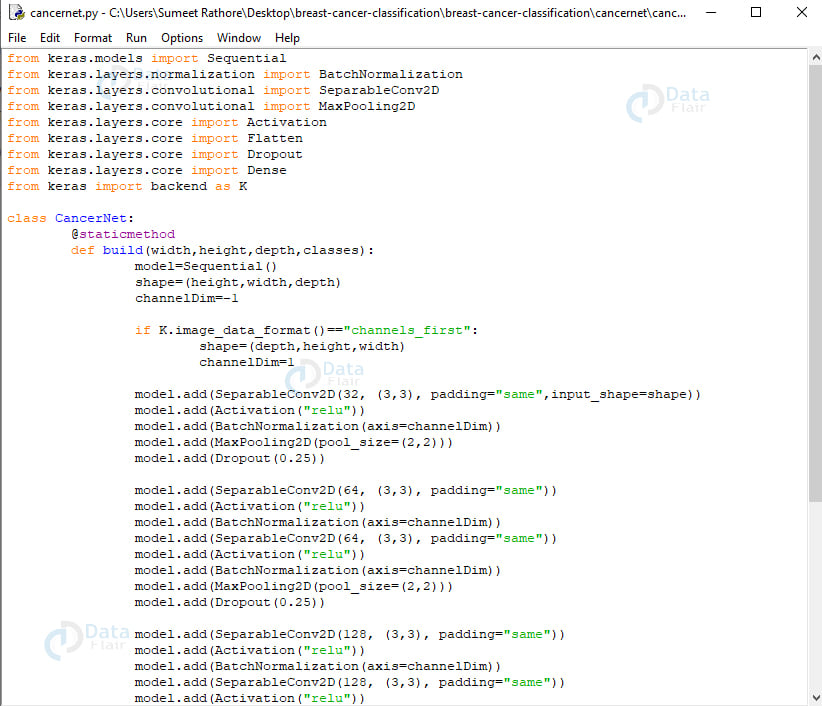
cancernet.py:
我们将构建的网络将是CNN(卷积神经网络),并将其称为CancerNet。该网络执行以下操作:
- 使用 3×3 个 CONV 滤波器
- 将这些过滤器堆叠在一起
- 执行最大池化
- 使用深度可分离卷积(效率更高,占用内存更少)
来自Keras。模型导入顺序来自Keras。层。规范化导入 BatchNormalization来自Keras。层。卷积导入 SeparableConv2D来自Keras。层。卷积导入 MaxPooling2D来自Keras。层。核心导入激活来自Keras。层。核心导入 Flatten来自Keras。层。核心导入 Dropout来自Keras。层。核心导入密集从 keras 导入后端为 K类 CancerNet:@staticmethoddef build(width,height,depth,classes):model=顺序()shape=(高度,宽度,深度)通道Dim=-1如果 K。image_data_format()==“channels_first”:shape=(深度,高度,宽度)通道Dim=1型。add(SeparableConv2D(32, (3,3), padding=“相同”,input_shape=形状))型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。添加(MaxPooling2D(pool_size=(2,2)))型。add(辍学(0.25))型。添加(SeparableConv2D(64, (3,3), padding=“相同”))型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。添加(SeparableConv2D(64, (3,3), padding=“相同”))型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。添加(MaxPooling2D(pool_size=(2,2)))型。add(辍学(0.25)型。add(SeparableConv2D(128, (3,3), padding=“相同”))
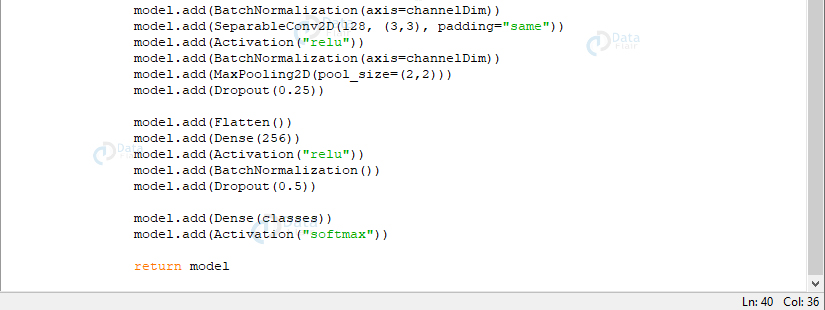
型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。add(SeparableConv2D(128, (3,3), padding=“相同”))型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。add(SeparableConv2D(128, (3,3), padding=“相同”))型。add(激活(“relu”))型。add(BatchNormalization(axis=channelDim))型。添加(MaxPooling2D(pool_size=(2,2)))型。add(辍学(0.25))型。add(扁平化())型。加(密集(256))型。add(激活(“relu”))型。add(BatchNormalization())型。add(辍学(0.5))型。add(Dense(类)))型。add(激活(“softmax”))返回模型
截图:
截图:
我们使用 Sequential API 来构建 CancerNet,并使用 SeparableConv2D 来实现深度卷积。类 CancerNet 有一个静态方法构建,它采用四个参数——图像的宽度和高度、深度(每张图像中的颜色通道数)以及网络将预测的类数,对我们来说,它是 2(0 和 1)。
在这种方法中,我们初始化模型和形状。使用 channels_first 时,我们会更新形状和通道尺寸。
现在,我们将定义三个 DEPTHWISE_CONV => RELU => POOL 层;每个都具有更高的堆叠和更多的过滤器。softmax 分类器输出每个类的预测百分比。最后,我们返回模型。
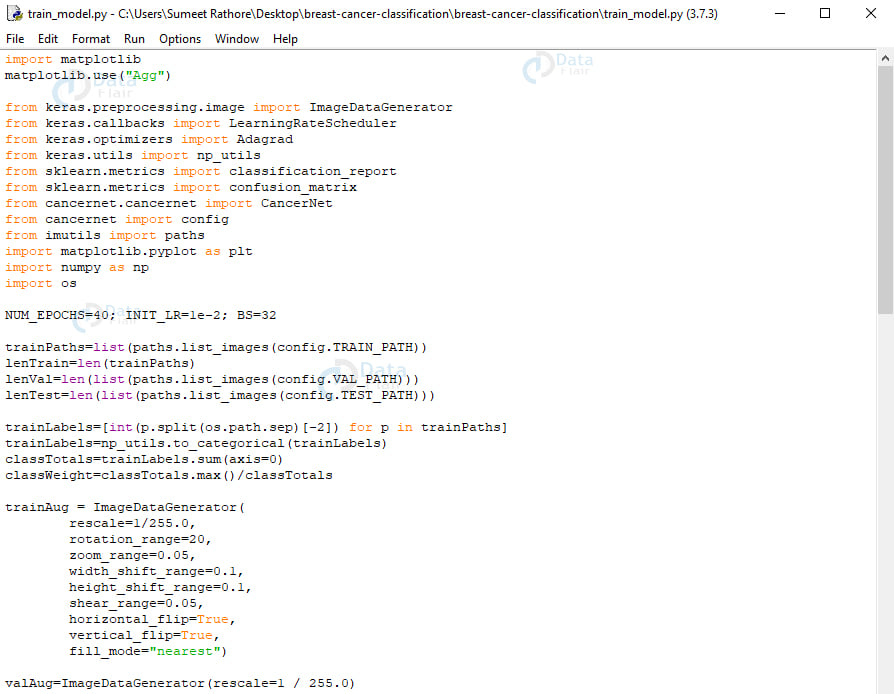
train_model.py:
这将训练和评估我们的模型。在这里,我们将从 keras、sklearn、cancernet、config、imutils、matplotlib、numpy 和 os 导入。
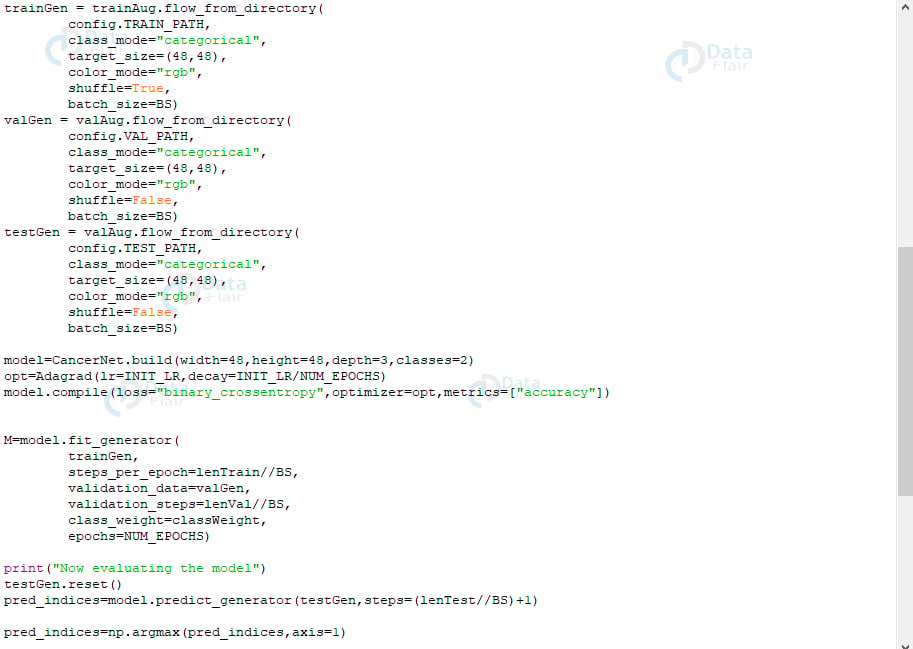
导入 matplotlibmatplotlib 中。use(“Agg”)来自Keras。预处理。图像导入 ImageDataGenerator来自Keras。回调导入 LearningRateScheduler来自Keras。optimizers import Adagrad来自Keras。utils 导入np_utils来自 Sklearn。指标导入classification_report来自 Sklearn。指标导入confusion_matrix来自 cancernet。cancernet 导入 CancerNet从 cancernet 导入配置从 imutils 导入路径导入 matplotlib。pyplot 作为 plt导入 numpy 作为 np导入操作系统NUM_EPOCHS=40;INIT_LR=1e-2;BS=32trainPaths=list(paths.list_images(config.TRAIN_PATH))lenTrain=len(trainPaths)lenVal=len(list(paths.list_images(config.VAL_PATH)))lenTest=len(list(paths.list_images(config.TEST_PATH)))trainLabels=[int(p.split(os.路径。sep)[-2]) for p in trainPaths]trainLabels=np_utils。to_categorical(trainLabels)classTotals=trainLabels。和(轴=0)classWeight=classTotals。max()/classTotalstrainAug = 图像数据生成器(重新缩放=1/255.0,rotation_range=20,zoom_range=0.05,width_shift_range=0.1,height_shift_range=0.1,shear_range=0.05,horizontal_flip=真,vertical_flip=真,fill_mode=“最
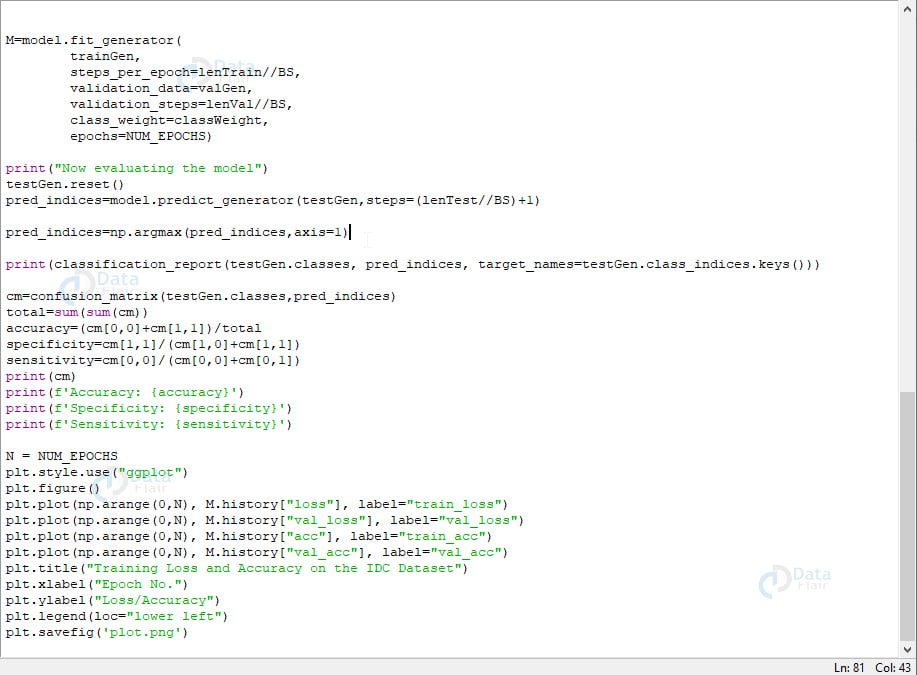
valAug=ImageDataGenerator(rescale=1 / 255.0)trainGen = 火车Aug。flow_from_directory(配置。TRAIN_PATH,class_mode=“分类”,target_size=(48,48),color_mode=“rgb”,shuffle=True,batch_size=BS)valGen = valAug。flow_from_directory(配置。VAL_PATH,class_mode=“分类”,target_size=(48,48),color_mode=“rgb”,shuffle=False,batch_size=BS)testGen = valAug。flow_from_directory(配置。TEST_PATH,class_mode=“分类”,target_size=(48,48),color_mode=“rgb”,shuffle=False,batch_size=BS)model=CancerNet。构建(宽度=48,高度=48,深度=3,类=2)opt=阿达格勒(lr=INIT_LR,衰变=INIT_LR/NUM_EPOCHS)型。compile(loss=“binary_crossentropy”,optimizer=opt,metrics=[“accuracy”])M=模型。fit_generator(trainGen,steps_per_epoch=lenTrainBS,validation_data=valGen,validation_steps=lenValBS,class_weight=classWeight,纪元=NUM_EPOCHS)print(“正在评估模型”)测试生成。重置()pred_indices=模型。predict_generator(testGen,steps=(lenTestBS)+1)pred_indices=np。argmax(pred_indices,轴=1)print(classification_report(testGen.类、pred_indices、target_names=testGen。class_indices。钥匙()))cm=confusion_matrix(testGen.类,pred_indices)总计=sum(sum(cm))
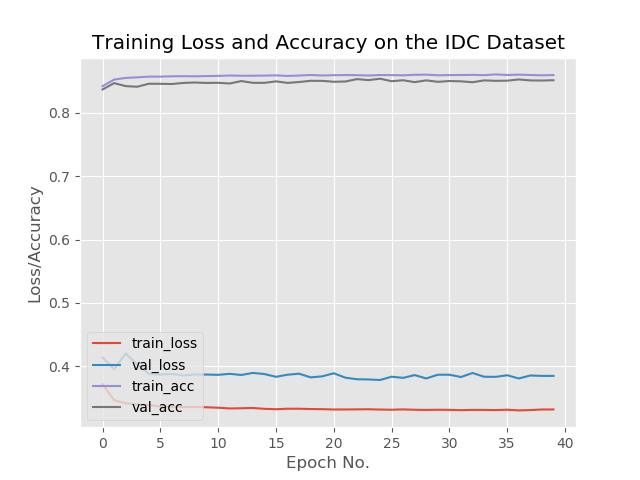
精度=(cm[0,0]+cm[1,1])/总计特异性=cm[1,1]/(cm[1,0]+cm[1,1])灵敏度=cm[0,0]/(cm[0,0]+cm[0,1])打印(cm)print(f'精度: {accuracy}')print(f'特异性: {特异性}')print(f'灵敏度: {sensitivity}')N = NUM_EPOCHSPLT。风格。use(“ggplot”)PLT。数字()PLT。plot(np.arange(0,N), M.history[“损失”], label=“train_loss”)PLT。plot(np.arange(0,N), M.历史[“val_loss”], label=“val_loss”)PLT。plot(np.arange(0,N), M.history[“acc”], label=“train_acc”)PLT。plot(np.arange(0,N), M.历史[“val_acc”], label=“val_acc”)PLT。title(“IDC 数据集上的训练损失和准确性”)PLT。xlabel(“纪元号”)PLT。ylabel(“损失/精度”)PLT。legend(loc=“左下角”)PLT。savefig('plot.png')
截图:
截图:
截图:
在此脚本中,首先,我们设置了 epoch 数、学习率和批处理大小的初始值。我们将获取三个目录中用于训练、验证和测试的路径数。然后,我们将获得训练数据的类权重,以便我们处理不平衡。
现在,我们初始化训练数据增强对象。这是一个有助于泛化模型的正则化过程。在这里,我们稍微修改了训练示例,以避免需要更多的训练数据。我们将初始化验证和测试数据增强对象。
我们将初始化训练、验证和测试生成器,以便它们可以生成大小为 batch_size 的批量图像。然后,我们将使用 Adagrad 优化器初始化模型,并使用 binary_crossentropy 损失函数对其进行编译。现在,为了拟合模型,我们调用 fit_generator()。
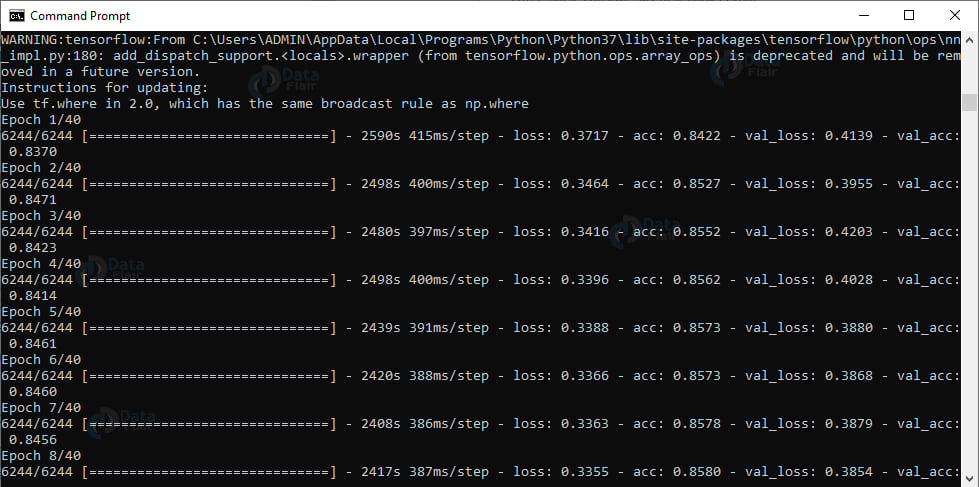
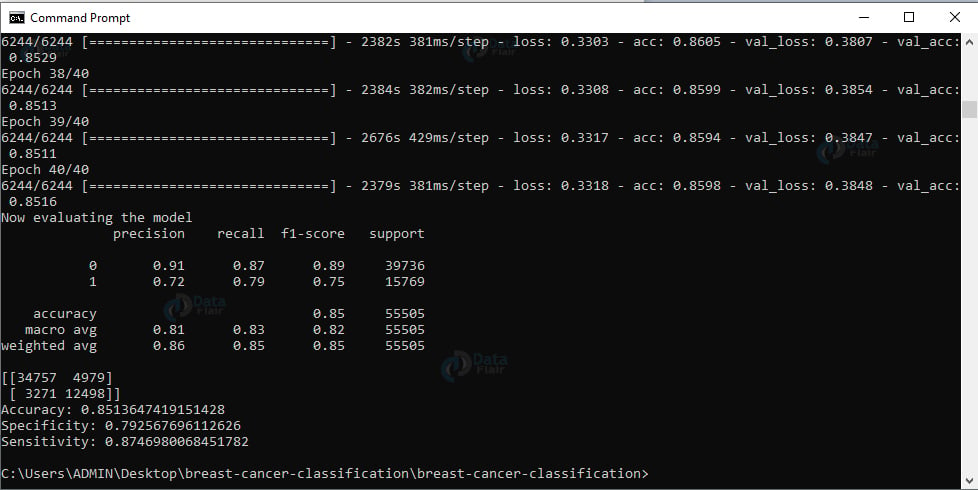
我们已经成功地训练了我们的模型。现在,让我们根据测试数据评估模型。我们将重置生成器并对数据进行预测。然后,对于来自测试集的图像,我们得到具有相应最大预测概率的标签的索引。我们将显示分类报告。
现在,我们将计算混淆矩阵并获得原始准确度、特异性和灵敏度,并显示所有值。最后,我们将绘制训练损失和准确性。
输出截图:
输出截图:
输出:
总结
在这个 python 项目中,我们学会了在 IDC 数据集上构建乳腺癌分类器(带有浸润性导管癌的组织学图像),并为其创建了网络 CancerNet。我们使用 Keras 来实现相同的结果。希望你喜欢这个 Python 项目。