单细胞数据的高维度常常导致“维度灾难”,使得细胞之间的关系难以准确揭示。传统的降维方法,如UMAP和tSNE,通常只能在低维空间中可视化数据,但会丢失重要的结构信息,特别是当数据存在复杂的非线性关系时。这些方法侧重于保留局部相似性,但忽略了细胞之间的全局关系。
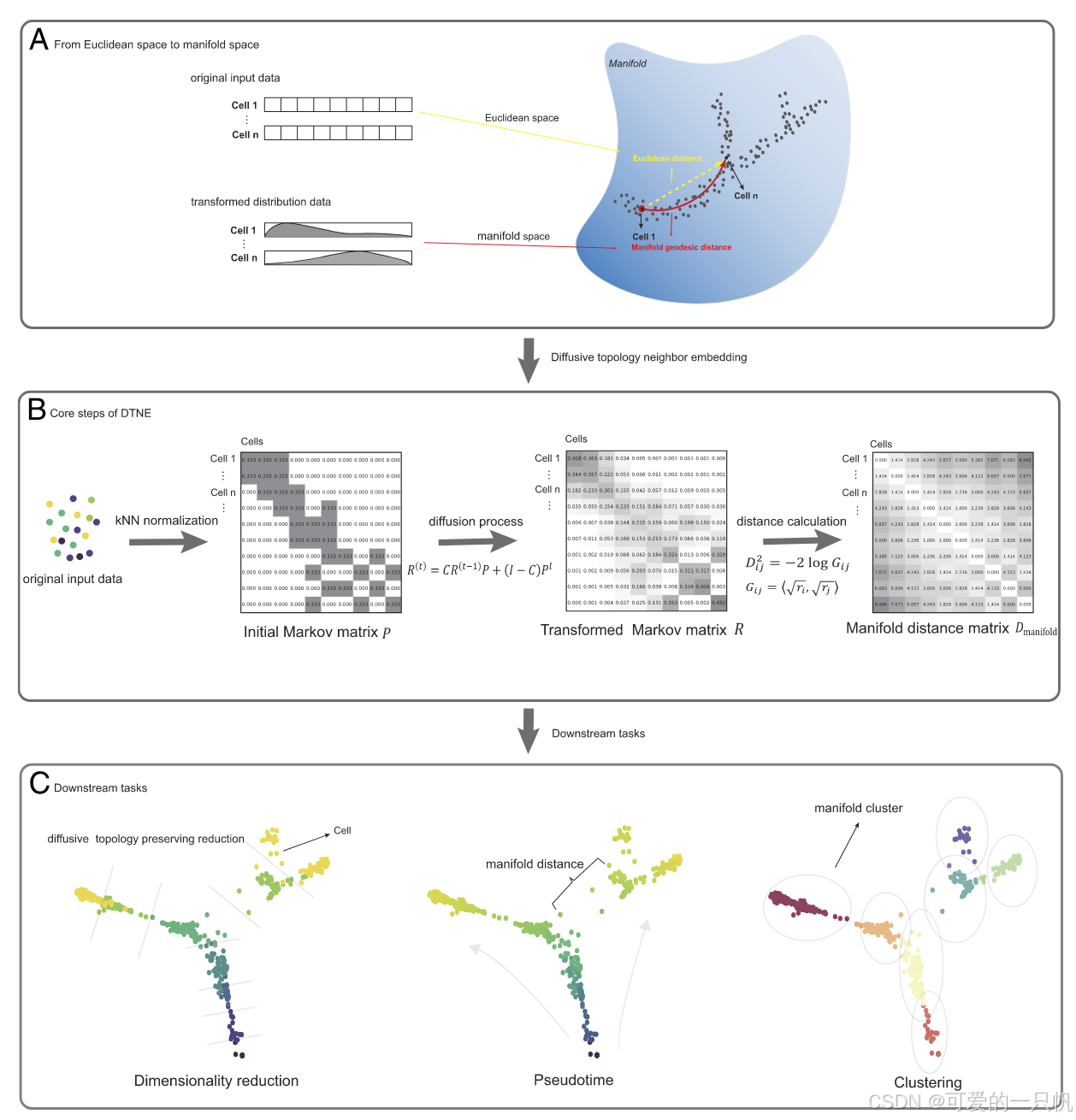
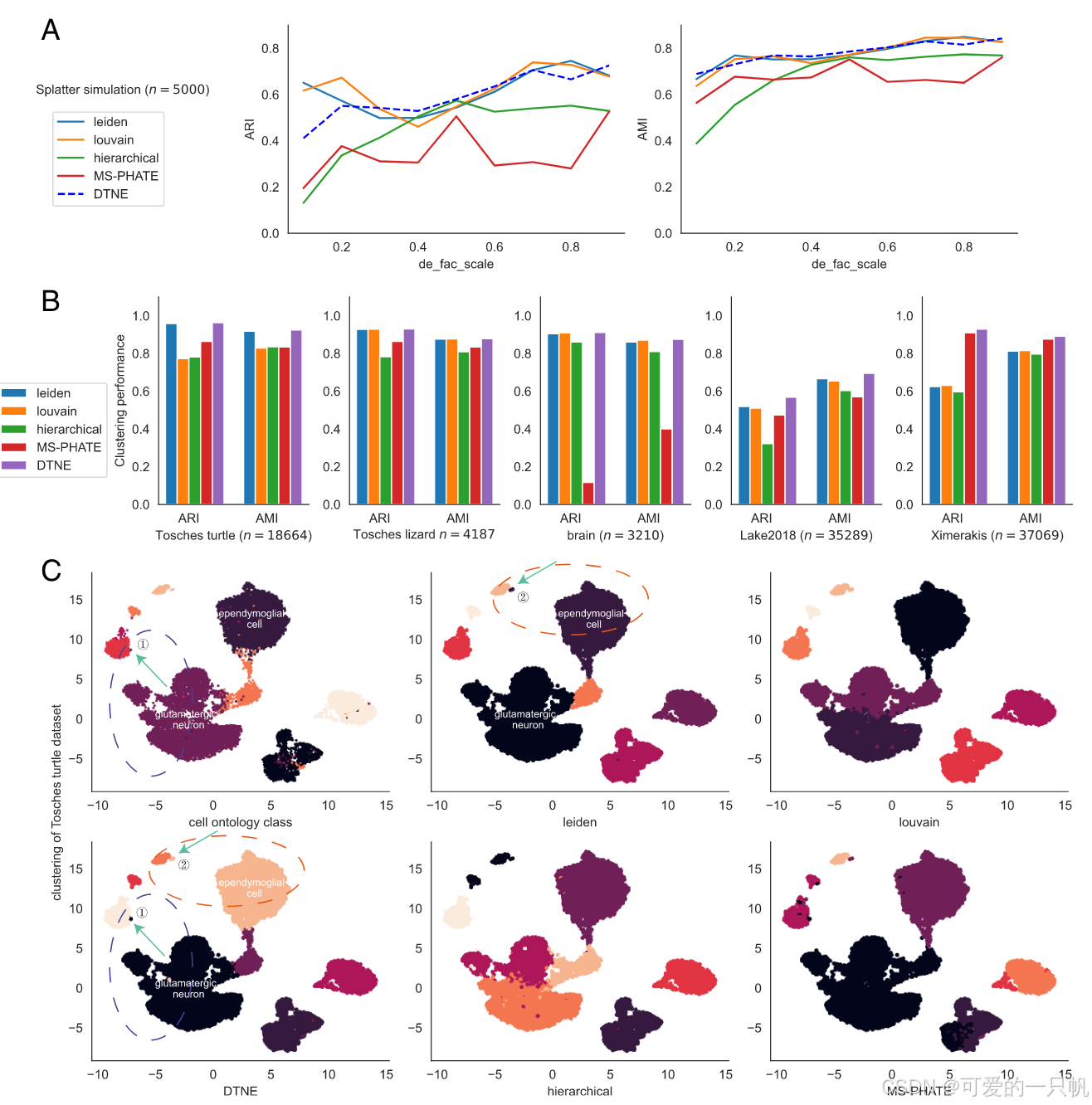
为了解决这一问题,DTNE(Diffusive Topology Neighbor Embedding)算法应运而生。DTNE通过模拟细胞间的扩散过程来构建流形距离矩阵,捕捉细胞间的真实拓扑关系。该算法不仅能够保留高维数据的地质距离,还能够进行降维、拟时序推断和细胞聚类分析,帮助研究者深入挖掘细胞之间的关系和动态变化。
算法原理:
核心思想:DTNE算法的核心是将欧几里得空间中的直线距离转化为流形空间中基于分布的地质距离,从而更准确地表示细胞之间的关系。
数据处理:首先,使用k近邻(kNN)算法将原始数据转化为细胞间的连接图,并归一化得到一个Markov矩阵。接着,通过个性化PageRank算法模拟图上的扩散动态,推导出一个扩散流形距离矩阵。这个矩阵不仅捕捉了细胞之间的局部关系,还考虑了全局的扩散模式。
分析任务:基于流形距离矩阵,DTNE可以执行关键的分析任务,如降维、拟时序推断和聚类分析,从而帮助研究者深入挖掘数据中的生物学信息。
参考文献:PNAS, 2025, 122(4), e2404860121.