实验四 基于字符串模式匹配算法的病毒感染检测
【实验目的】
1.掌握字符串的顺序存储表示方法。
2.掌握字符串模式匹配BF算法和KMP算法的实现。
【实验内容】
问题描述
医学研究者最近发现了某些新病毒,通过对这些病毒的分析,得知它们的DNA序列都是环状的。现在研究者已收集了大量的病毒DNA和人的DNA数据,想快速检测出这些人是否感染了相应的病毒。为了方便研究,研究者将人的DNA和病毒DNA均表示成由一些字母组成的字符序列,然后检测某种病毒DNA序列是否在患者的DNA序列中出现过。如果出现过,则此人感染了该病毒,否则没有感染。例如,假设病毒的DNA序列为baa,患者1的DNA序列为aaabbba,则感染;患者2的DNA序列为babbba,则未感染。(注意,人的DNA序列是线性的,而病毒的DNA序列是环状的。)
输入要求
多组数据,每组数据有1行,为序列A和B,A对应病毒的DNA序列,B对应人的DNA序列。A和B都为“0”时输入结束。
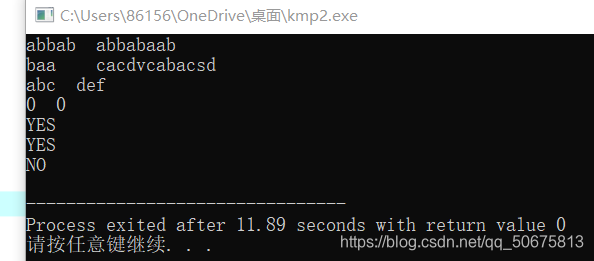
输入样例
abbab abbabaab
baa cacdvcabacsd
abc def
0 0
输出样例
YES
YES
NO
【实验提示】
此实验内容即要求实现教材算法的具体案例。利用BF算法来实现字符串的模式匹配过程的,效率较低,。利用KMP算法完成模式匹配以提高算法的效率。
解决方法1:暴力算法
#include <stdio.h> // printf(); scanf()
#include <stdlib.h> // exit()
#include <malloc.h> // malloc()
#include <time.h> // srand((unsigned)time(NULL));
#include <string.h>
#define TRUE 1
#define FALSE 0
#define OK 1
#define ERROR 0
typedef int Status;
typedef int ElemType;
#define MAXSTRLEN 255
typedef unsigned char SString[MAXSTRLEN + 1];
Status StrAssign(SString &T, char *chars) {
if(strlen(chars) > MAXSTRLEN)
return ERROR;
else {
T[0] = strlen(chars);
for(int i=1; i<=T[0]; i++)
T[i] = *(chars+i-1);
return OK;
}
}
Status StrCopy(SString &T, SString S) {
for(int i=0; i<=S[0]; i++)
T[i] = S[i];
return OK;
}
int StrLength(SString S) {
return S[0];
}
int index(SString S,SString T,int pos)
{
int i=pos,j=1;
while(i<=S[0]&&j<=T[0])
{
if(S[i]==T[j])
{
++i;
++j;
}
else
{
i=i-j+2;
j=1;
}
}
if(j>T[0])
return i-T[0];
else return 0;
}
int main()
{
int flag[100];
int w=0;
int x;int number;int pos;
char c[MAXSTRLEN+1],d[MAXSTRLEN+1];
SString S;
SString T;
SString Z;
do
{
scanf("%s",c);
scanf("%s",d);
StrAssign(S, c);
StrAssign(T, d);
char str[T[0]];
for(int i=0; i<=S[0]; i++)
str[i] = T[i+1];
int i=0;
Z[0]=T[0];
for(i=0;i<T[0];i++)
{
for(int j=1;j<=T[0];j++)
{
Z[j]=str[(j+i)%(T[0])];
}
number=index(S,Z,pos);
if(number!=0&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
{
flag[w++]=1;
break;
}
}
if(i==T[0]&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
flag[w++]=0;
}while(strcmp(c,"0")!=0&&strcmp(d,"0")!=0);
for(int e=0;e<w;e++)
{
if(flag[e]==1)
printf("YES\n");//即在人体dna中找到与病毒dna相同的序列
else
printf("NO\n");//即在人体dna中找到与病毒dna相同的序列
}
return 0;
}
解决方法2:kmp算法
#include <stdio.h> // printf(); scanf()
#include <stdlib.h> // exit()
#include <malloc.h> // malloc()
#include <time.h> // srand((unsigned)time(NULL));
#include <string.h>
#define TRUE 1
#define FALSE 0
#define OK 1
#define ERROR 0
typedef int Status;
typedef int ElemType;
#define MAXSTRLEN 255
typedef unsigned char SString[MAXSTRLEN + 1];
Status StrAssign(SString &T, char *chars) {
if(strlen(chars) > MAXSTRLEN)
return ERROR;
else {
T[0] = strlen(chars); // 0号单元存放串的长度
for(int i=1; i<=T[0]; i++)
T[i] = *(chars+i-1);
return OK;
}
}
Status StrCopy(SString &T, SString S) {
for(int i=0; i<=S[0]; i++)
T[i] = S[i];
return OK;
}
int StrLength(SString S) {
return S[0];
}
void get_next(SString T,int next[])
{
int j;
int i=1;next[1]=0;j=0;
while(i<T[0])
{
if(j==0||T[i]==T[j]){i++;j++;next[i]=j;}
else j=next[j];
}
}
int Index_KMP(SString S,SString T,int pos)
{
int next[MAXSTRLEN+1];
get_next(T,next);
int i=pos; int j=1;
while(i<=S[0]&&j<=T[0])//i j都不超过其串的长度
{//失配
//1:失配,当j==0时,则目标主串的检测指针前进一位,模式串检测指针回到T[1].进行下一趟的比较
//2:失配,当j>0时,那么在下一趟比较时,模式串的起始位置为Tnext[j],目标主串S的检测指针不回溯,仍然指向上一趟失配的位置
if(j==0||S[i]==T[j])
{
++i;++j;//继续比较后继字符
}
else j=next[j];//模式串向右移动
}
if(j>T[0]) return i-T[0];//匹配成功
else return 0;
}
int main()
{
int flag[100];
int w=0;
int x;int number;int pos;
char c[MAXSTRLEN+1],d[MAXSTRLEN+1];
SString S;
SString T;
SString Z;
do
{
scanf("%s",d);
scanf("%s",c);
StrAssign(S, c);
StrAssign(T, d);
char str[T[0]];
for(int i=0; i<=S[0]; i++)
str[i] = T[i+1];
int i=0;
Z[0]=T[0];
for(i=0;i<T[0];i++)
{
for(int j=1;j<=T[0];j++)
{
Z[j]=str[(j+i)%(T[0])];
}
//StrPrint(Z);
//printf("\n");
number=Index_KMP(S,Z,pos);
if(number!=0&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
{
flag[w++]=1;
break;
}
}
if(i==T[0]&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
flag[w++]=0;
}while(strcmp(c,"0")!=0&&strcmp(d,"0")!=0);
for(int e=0;e<w;e++)
{
if(flag[e]==1)
printf("YES\n");//即在人体dna中找到与病毒dna相同的序列
else
printf("NO\n");//即在人体dna中找到与病毒dna相同的序列
}
return 0;
}
解决方法3:kmp算法改进版
```c
#include <stdio.h> // printf(); scanf()
#include <stdlib.h> // exit()
#include <malloc.h> // malloc()
#include <time.h> // srand((unsigned)time(NULL));
#include <string.h>
#define TRUE 1
#define FALSE 0
#define OK 1
#define ERROR 0
typedef int Status;
typedef int ElemType;
#define MAXSTRLEN 255
typedef unsigned char SString[MAXSTRLEN + 1];
Status StrAssign(SString &T, char *chars) {
if(strlen(chars) > MAXSTRLEN)
return ERROR;
else {
T[0] = strlen(chars);
for(int i=1; i<=T[0]; i++)
T[i] = *(chars+i-1);
return OK;
}
}
int StrLength(SString S) {
return S[0];
}
void get_nextval(SString T,int nextval[])
{
int j;
int i=1;nextval[1]=0;j=0;
while(i<T[0])
{
if(j==0||T[i]==T[j])
{i++;j++;
if(T[i]!=T[j])
nextval[i]=j;
else
nextval[i]=nextval[j];
}
else j=nextval[j];
}
}
int Index_KMP_val(SString S,SString T,int pos)
{
int nextval[MAXSTRLEN+1];
get_nextval(T,nextval);
int i=pos; int j=1;
while(i<=S[0]&&j<=T[0])//i j都不超过其串的长度
{//失配
//1:失配,当j==0时,则目标主串的检测指针前进一位,模式串检测指针回到T[1].进行下一趟的比较
//2:失配,当j>0时,那么在下一趟比较时,模式串的起始位置为Tnext[j],目标主串S的检测指针不回溯,仍然指向上一趟失配的位置
if(j==0||S[i]==T[j])
{
++i;++j;//继续比较后继字符
}
else j=nextval[j];//模式串向右移动
}
if(j>T[0]) return i-T[0];//匹配成功
else return 0;
}
int main()
{
int flag[100];//用于计算循环数组 每次的结果
int w=0; //用于统计flag数组
int x;int number;int pos;
char c[MAXSTRLEN+1],d[MAXSTRLEN+1];
SString S;
SString T;
SString Z;
do
{
scanf("%s",d);
scanf("%s",c);
StrAssign(S, c);
StrAssign(T, d);
char str[T[0]];
for(int i=0; i<=S[0]; i++)
str[i] = T[i+1];
int i=0;
Z[0]=T[0];
for(i=0;i<T[0];i++)
{
for(int j=1;j<=T[0];j++)
{
Z[j]=str[(j+i)%(T[0])];
}
number=Index_KMP_val(S,Z,pos);
if(number!=0&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
{
flag[w++]=1;
break;
}
}
if(i==T[0]&&strcmp(c,"0")!=0&&strcmp(d,"0")!=0)
flag[w++]=0;
}while(strcmp(c,"0")!=0&&strcmp(d,"0")!=0);
for(int e=0;e<w;e++)
{
if(flag[e]==1)
printf("YES\n");//即在人体dna中找到与病毒dna相同的序列
else
printf("NO\n");//即在人体dna中找到与病毒dna相同的序列
}
return 0;
}