转录因子(TF)是通过结合 DNA 序列中的转录因子结合位点(TFBS)来调控基因转录所必需的蛋白质。准确预测 TFBS 有助于设计和构建基于 TF 的代谢调控系统。尽管已经开发了各种用于预测 TFBS 的深度学习算法,但预测性能仍有待提高。本文提出了一种基于 Transformer 的双向编码器表示 (BERT) 的模型 BERT-TFBS,用于仅基于 DNA 序列预测 TFBS。该模型由预训练的 BERT 模块(DNABERT-2)、卷积神经网络 (CNN) 模块、卷积块注意模块 (CBAM) 和输出模块组成。BERT-TFBS 模型利用预训练的 DNABERT-2 模块通过迁移学习方法获取 DNA 序列中复杂的长期依赖关系,并应用 CNN 模块和 CBAM 提取高阶局部特征。所提出的模型基于 165 个 ENCODE ChIP-seq 数据集进行训练和测试。我们对模型变体、跨细胞系验证和与其他模型的比较进行了实验。实验结果证明了 BERT-TFBS 在预测 TFBS 方面的有效性和泛化能力,并表明所提出的模型优于其他深度学习模型。BERT-TFBS 的源代码可在https://github.com/ZX1998-12/BERT-TFBS上找到。
1.总体框架
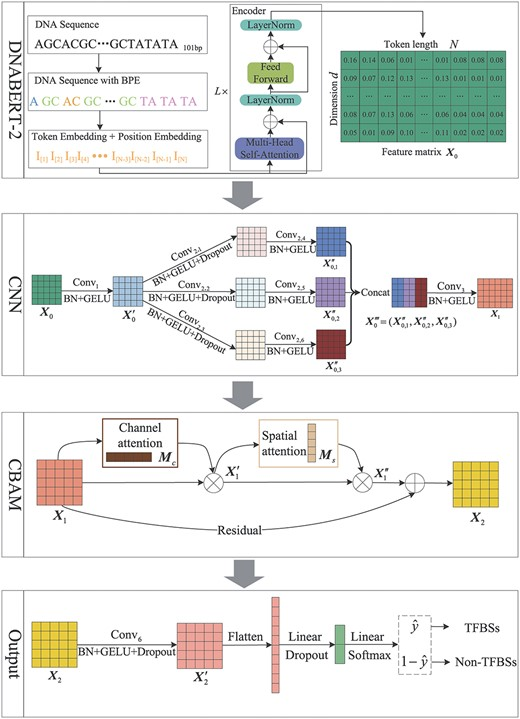
所提出的 BERT-TFBS 的整体框架如图 1所示。该模型由四个模块组成:预训练的 DNABERT-2 模块、CNN 模块、CBAM 和输出模块。BERT-TFBS 中卷积操作的详细信息在补充表 S2。
DNABERT-2:该模块是一个预先训练的 BERT 模型,用于编码 DNA 序列并提取序列内的长期依赖关系。
CNN模块:该模块通过一系列卷积层提取序列矩阵的高阶局部特征。
CBAM:该模块用于通过空间和通道注意机制增强局部特征。
输出模块:该模块整合获取的序列特征,采用多层感知器提供识别 DNA 序列中 TFBS 的输出结果。
基于模型架构和交叉熵损失函数,BERT-TFBS 的训练过程总结如算法 1 所示。所提出的模型可以使用 PyTorch 1.12.0 实现和训练,小批量大小为 32。超参数值如下:补充表 S3。在训练过程中,利用 AdamW 优化算法 根据损失函数的下降梯度优化模型参数。此外,使用预热 和余弦退火技术 调整优化器的学习率。总训练周期数设置为 15,其中预热期有 5 个周期,余弦退火阶段有 10 个周期。为了防止 BERT 模型在微调过程中不稳定,选择了相对较低的学习率。在预热阶段,学习率系统地从较小的值上升到 1.5e-5。在余弦退火阶段,学习率根据余弦函数系统地减小到 2e-6。为了避免过度拟合,我们应用了 dropout 操作。因此,输出模块中的dropout rate设置为0.4和0.5,但由于CNN模块的结构更复杂,因此CNN模块的dropout rate设置为0.2,以增加模型的正则化。