介绍
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)聚类算法,它是一种基于高密度连通区域的基于密度的聚类算法,能够将具有足够高密度的区域划分为簇,并在具有噪声的数据中发现任意形状的簇。
DBSCAN需要两个重要参数:epsilon(eps)和最小点(minPts)。参数eps定义了点x附近的邻域半径ε,它被称为x的最邻居。参数minPts是eps半径内的最小邻居数。
上图中(a),数据集中的任何点x邻居(6=minPts)都被标记为核心点,ε是半径。上图中(b),x为核心点,y的邻居小于(4<minpts)是边界点,但它属于核心点x的最邻居。z点既不是核心也不是边界点,它被称为噪声点或异常值。
dbscan算法将数据点分为三类:
核心点:在半径eps内含有超过minPts数目的点。
边界点:在半径eps内点的数量小于使用DBSCAN进行聚类的时候,不需要预先指定簇的个数,最终的簇的个数不确定。minPts,但是落在核心点的邻域内的点。
噪音点:既不是核心点也不是边界点的点
DBSCAN算法的执行过程
1、DBSCAN算法随机从一个未被访问的数据点x开始,以eps为半径搜索范围内的所有邻域点。
2、如果x点在该邻域内有足够数量的点,数量大于等于minPts,则聚类过程开始,并且当前数据点成为新簇中的第一个核心点。否则,该点将被标记为噪声。该点都会被标记为“已访问”。
3、新簇中的每个核心点x,它的eps距离邻域内的点会归为同簇。eps邻域内的所有点都属于同一个簇,然后对才添加到簇中的所有新点重复上述过程。
4、重复步骤2和3两个过程,直到确定了簇中的所有点才停止,即访问和标记了聚类的eps邻域内的所有点。
5、当完成了这个簇的划分,就开始处理新的未访问的点,发现新的簇或者是噪声。重复上述过程,直到所有点被标记为已访问才停止。这样就完成了,对所有点的聚类过程。
优点和缺点
DBSCAN具有很多优点,提前不需要确定簇的数量。不同于Mean-shift算法,当数据点非常不同时,会将它们单纯地引入簇中,DBSCAN能将异常值识别为噪声。另外,它能够很好地找到任意大小和任意形状的簇。
DBSCAN算法的主要缺点是,当数据簇密度不均匀时,它的效果不如其他算法好。这是因为当密度变化时,用于识别邻近点的距离阈值ε和minPoints的设置将随着簇而变化。在处理高维数据时也会出现这种缺点,因为难以估计距离阈值eps。
dbscan包介绍
dbscan包,提供了基于密度的有噪声聚类算法的快速实现,包括 DBSCAN(基于密度的具有噪声的应用的空间聚类),OPTICS(用于识别聚类结构的排序点),HDBSCAN(分层DBSCAN)和LOF(局部异常因子)算法,dbscan底层使用C++编程,并建立kd树的数据结构进行更快的K最近邻搜索,从而实现加速。
dbscan包的安装非常简单,只需要一条命令就能完成。
install.packages("dbscan")
library(dbscan)
函数列表:
dbscan(), 实现DBSCAN算法
optics(), 实现OPTICS算法
hdbscan(), 实现带层次DBSCAN算法
sNNclust(), 实现共享聚类算法
jpclust(), Jarvis-Patrick聚类算法
lof(), 局部异常因子得分算法
extractFOSC(),集群优选框架,可以通过参数化来执行聚类。
frNN(), 找到固定半径最近的邻居
kNN(), 最近邻算法,找到最近的k个邻居
sNN(), 找到最近的共享邻居数量
pointdensity(), 计算每个数据点的局部密度
kNNdist(),计算最近的k个邻居的距离
kNNdistplot(),画图,最近距离
hullplot(), 画图,集群的凸壳
kNN()函数,使用kd-tree数据结构,用来快速查找数据集中的所有k个最近邻居。
kNN()函数的介绍
函数定义:
kNN(x, k, sort = TRUE, search = "kdtree", bucketSize = 10, splitRule = "suggest", approx = 0)
参数列表
x,数据矩阵,dist对象或kNN对象。
k,要查找的邻居数量。
sort,按距离对邻居进行排序。
search,最近邻搜索策略,使用kdtree,linear或dist三选一,默认为kdtree。
bucketSize,kd-tree叶子节点的最大值。
splitRule,kd-tree的拆分规则,默认用SUGGEST。
approx,使用近似方法,加速计算。
函数使用:
以iris鸢尾花的数据集,做为样本。聚类是不需要有事前有定义的,所以我们把iris的种属列去掉。
去掉种属列
iris2 <- iris[, -5]
head(iris2)
Sepal.Length Sepal.Width Petal.Length Petal.Width
1 5.1 3.5 1.4 0.2
2 4.9 3.0 1.4 0.2
3 4.7 3.2 1.3 0.2
4 4.6 3.1 1.5 0.2
5 5.0 3.6 1.4 0.2
6 5.4 3.9 1.7 0.4
使用kNN()函数,来计算iris2数据集中,每个值最近的5个点。
查询最近邻的5个点
nn <- kNN(iris2, k=5)
打印nn对象
nn
k-nearest neighbors for 150 objects (k=5).
Available fields: dist, id, k, sort
查询nn的属性列表
attributes(nn)
$`names`
[1] "dist" "id" "k" "sort"
$class
[1] "kNN" "NN"
打印出,每个点最近邻的5个点。行,为每个点索引值,列,为最近邻的5个点,输出的矩阵为索引值。
head(nn$id)
1 2 3 4 5
[1,] 18 5 40 28 29
[2,] 35 46 13 10 26
[3,] 48 4 7 13 46
[4,] 48 30 31 3 46
[5,] 38 1 18 41 8
[6,] 19 11 49 45 20
打印出,每个点与最近的5个点的距离值。行,为每个点的索引,列,为最近邻的5个点,输出的矩阵为距离值。
head(nn$dist)
1 2 3 4 5
[1,] 0.1000000 0.1414214 0.1414214 0.1414214 0.1414214
[2,] 0.1414214 0.1414214 0.1414214 0.1732051 0.2236068
[3,] 0.1414214 0.2449490 0.2645751 0.2645751 0.2645751
[4,] 0.1414214 0.1732051 0.2236068 0.2449490 0.2645751
[5,] 0.1414214 0.1414214 0.1732051 0.1732051 0.2236068
[6,] 0.3316625 0.3464102 0.3605551 0.3741657 0.3872983
如果我们要查看索引为33的点,与哪5个点最紧邻,可以用下面的方法。
#设置索引
idx<-33
# 打印与33,最近邻的5个点的索引
nn$id[idx,]
1 2 3 4 5
34 47 20 49 11
# 画图
cols = ifelse(1:nrow(iris2) %in% nn$id[idx,],"red", "black")
cols[idx]<-'blue'
plot(iris2,pch = 19, col = cols)
我们的数据集是多列的,把每2列组合形成的二维平面,都进行输出。蓝色表示索引为33的点,红色表示最紧邻的5个点,黑色表示其他的点。
从图中,可以很直观的看到,这几点确实是密集的在一起,也就是找到了最近邻。
接下来,我们画出连线图,选取第一列(Sepal.Length)和第二列(Sepal.Width),按取画出最紧邻前5连接路径。
plot(nn, iris2)
通过连接路径,我们就能很清晰的看到,最紧邻算法的分组过程,连接在一起的就够成了一个分组,没有连接在一起的就是另外的分组,上图中可以看出来分成了2个组。
再对nn进行二次最近邻计算,画出前2的连接路径。
plot(kNN(nn, k = 2), iris2)
dbscan()函数使用
dbscan是一种基于密度的聚类算法,这类密度聚类算法一般假定类别可以通过样本分布的紧密程度决定。同一类别的样本,他们之间的紧密相连的,也就是说,在该类别任意样本周围不远处一定有同类别的样本存在。
函数定义:
dbscan(x, eps, minPts = 5, weights = NULL, borderPoints = TRUE, ...)
参数解释:
x, 矩阵或者距离对象,frNN对象。
eps,半径的大小。
minPts, 半径区域中的最小点数量,默认为5
weights, 数据点的权重,仅用于加权聚类
borderPoints,边界点是否为噪声,默认为TRUE;为FALSE时,边界点为噪声。
…,将附加参数传递给固定半径最近邻搜索算法,调用frNN。
函数使用:
以iris鸢尾花的数据集,做为样本。聚类是不需要有事前有定义的,所以我们把iris的种属列去掉。
# 去掉种属列
iris2 <- iris[, -5]
head(iris2)
Sepal.Length Sepal.Width Petal.Length Petal.Width
1 5.1 3.5 1.4 0.2
2 4.9 3.0 1.4 0.2
3 4.7 3.2 1.3 0.2
4 4.6 3.1 1.5 0.2
5 5.0 3.6 1.4 0.2
6 5.4 3.9 1.7 0.4
在使用dbscan函数时,我们要输出2个参数,eps和minPts。
eps,值可以使用绘制k-距离曲线(k-distance graph)方法得到,在k-距离曲线图明显拐点位置为较好的参数。若参数设置过小,大部分数据不能聚类;若参数设置过大,多个簇和大部分对象会归并到同一个簇中。
minPts,通常让minPts≥dim+1,其中dim表示数据集聚类数据的维度。若该值选取过小,则稀疏簇中结果由于密度小于minPts,从而被认为是边界点儿不被用于在类的进一步扩展;若该值过大,则密度较大的两个邻近簇可能被合并为同一簇。
下面我们通过绘制k-距离曲线,寻找knee,即明显拐点位置为对应较好的参数,找到适合的eps值。使用kNNdistplot()函数,让参数k=dim + 1,dim为数据集列的个数,iris2是4列,那么设置k=5。
# 画出最近距离图
kNNdistplot(iris2, k = 5)
abline(h=0.5, col = "red", lty=2)
kNNdistplot()会计算点矩阵中的k=5的最近邻的距离,然后按距离从小到大排序后,以图形进行展示。x轴为距离的序号,y轴为距离的值。图中黑色的线,从左到右y值越来越大。
通过人眼识别,k-距离曲线上有明显拐点,我们以y=0.5平行于x轴画一条红色线,突出标识。所以,最后确认的eps为0.5。
调用dbscan()函数,进行对iris2数据集进行聚类,eps=0.5,minPts=5。
res <- dbscan(iris2, eps = 0.5, minPts = 5)
res
DBSCAN clustering for 150 objects.
Parameters: eps = 0.5, minPts = 5
The clustering contains 2 cluster(s) and 17 noise points.
0 1 2
17 49 84
Available fields: cluster, eps, minPts
聚类后,一共分成了2组,第1组49个值,第2组84个值,另外,第0组17个值为噪声点。把聚类的结果画图展示。
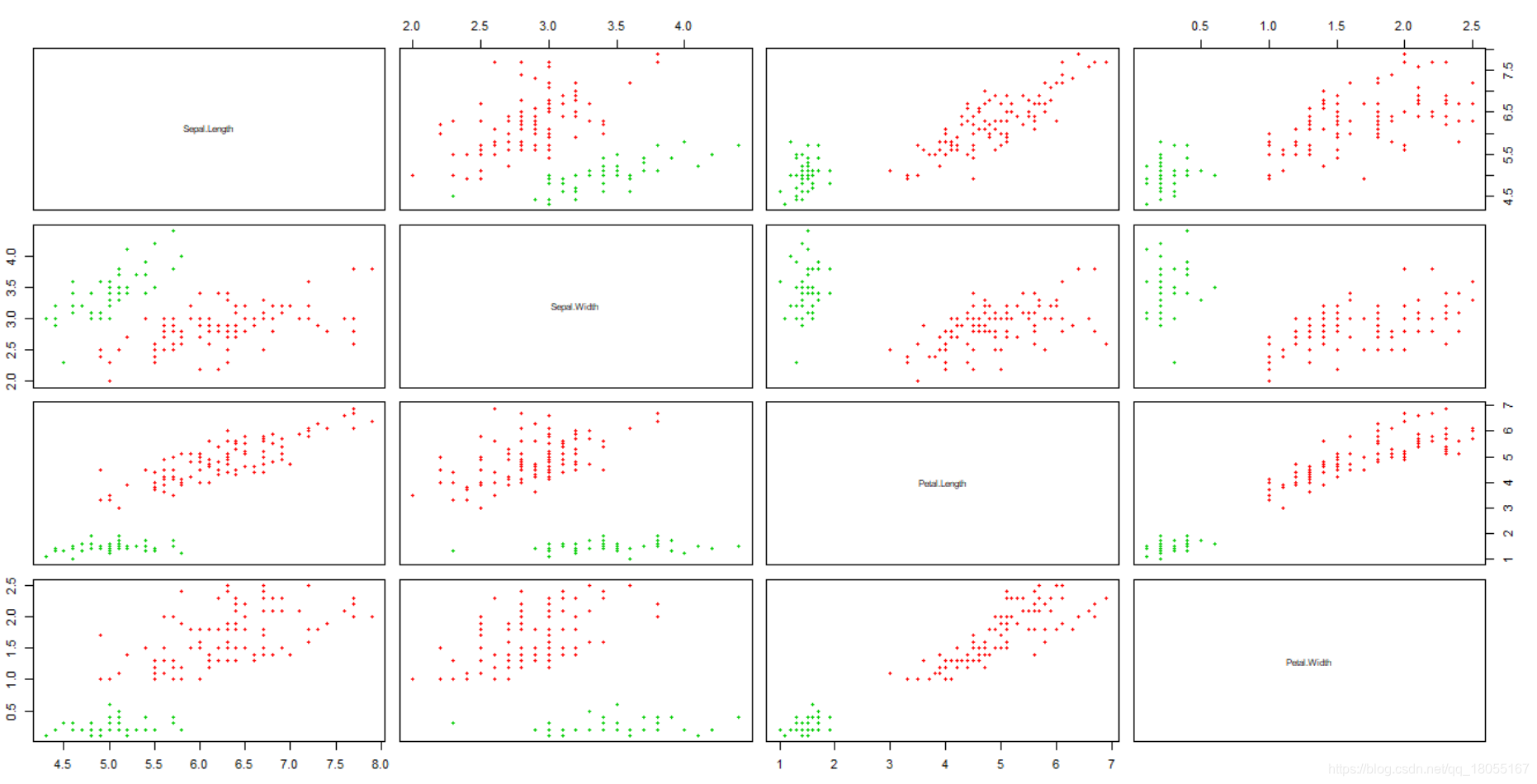
pairs(iris, col = res$cluster + 1L)
数据集是多列的,把每2列组合形成的二维平面,都进行输出。红色点表示第1组,绿色点表示为第2组,黑色点表示噪声点。这样就完成了有噪声的基于密度的dbscan聚类。
hdbscan()函数使用
hdbscan(),快速实现了分层DBSCAN算法,与stats包中的hclust()方法形成的传统分层聚类方法类似。
函数定义:
hdbscan(x, minPts, xdist = NULL,gen_hdbscan_tree = FALSE, gen_simplified_tree = FALSE)
参数解释:
x,矩阵或者距离对象
minPts,区域中的最小点数量
xdist,dist对象,可以提前算出来,当参数传入
gen_hdbscan_tree,生成一个hdbscan树
gen_simplified_tree,生成一个简化的树结构
iris鸢尾花的数据集
以iris鸢尾花的数据集,做为样本,去掉种属列。设置minPts =5让当前群集中最小的数量为5,开始聚类。
hcl<-hdbscan(iris2, minPts = 5)
hcl
HDBSCAN clustering for 150 objects.
Parameters: minPts = 5
The clustering contains 2 cluster(s) and 0 noise points.
1 2
100 50
Available fields: cluster, minPts, cluster_scores, membership_prob,
outlier_scores, hc
聚类后,一共分成了2组,第1组100个值,第2组50个值,没有噪声点。生成的hcl对象包括6个属性。
属性解释
cluster,表明属性哪个群集,零表示噪声点。
minPts,群集中最小的数量
cluster_scores,每个突出(“平坦”)群集的稳定性分数之和。
membership_prob,群集内某点的“概率”或个体稳定性
outlier_scores,每个点的异常值
hc,层次结构对象
把聚类的结果画图展示
plot(iris2, col=hcl$cluster+1, pch=20)
数据集是多列的,把每2列组合形成的二维平面,都进行输出。红色点表示第1组,绿色点表示为第2组,这样就完成了hdbscan聚类。
打印hcl对象层次结构,包括150个数据,聚法方法是健壮单一的,距离是相互可达。
hcl$hc
Call:
hdbscan(x = iris2, minPts = 5)
Cluster method : robust single
Distance : mutual reachability
Number of objects: 150
画出层次的合并过程图
plot(hcl$hc, main="HDBSCAN* Hierarchy")
从图可以清楚的看出,主要的2类的分支,区分度比较高。
** moons数据集**
由于iris数据集用hdbscan聚类获得的结果,与真实的数据分类结果不一致。我们再用dbscan包自带的数据集moons做一下测试。
先准备数据,加载moons数据集,了解数据基本情况,画出散点图。
# 加载dbscan自带数据集
data("moons")
head(moons)
X Y
1 -0.41520756 1.0357347
2 0.05878098 0.3043343
3 1.10937860 -0.5097378
4 1.54094828 -0.4275496
5 0.92909498 -0.5323878
6 -0.86932470 0.5471548
# 画出散点图
plot(moons, pch=20)
用hdbscan()函数,实现层次dbscan算法。
cl <- hdbscan(moons, minPts = 5)
cl
HDBSCAN clustering for 100 objects.
Parameters: minPts = 5
The clustering contains 3 cluster(s) and 0 noise points.
1 2 3
25 25 50
Available fields: cluster, minPts, cluster_scores, membership_prob,
outlier_scores, hc
一共100条数据,被分成了3类,没有噪声。把聚类的结果画图展示。
# 画图
plot(moons, col=cl$cluster+1, pch=20)
打印层次结构
cl$hc
Call:
hdbscan(x = moons, minPts = 5)
Cluster method : robust single
Distance : mutual reachability
Number of objects: 100
画出层次的合并过程图
plot(cl$hc, main="HDBSCAN* Hierarchy")
从图可以清楚的看出,主要的3类的分支,区分度比较高。
如果我们想省略分层的细节,我们可以只画出主要分支,并标识类别。
plot(cl, gradient = c("purple", "blue", "green", "yellow"), show_flat = T)
接下来,我们要对群集的稳定性做一些优化,cluster_scores属性可以查看集群的得分。
cl$cluster_scores
1 2 3
110.70613 90.86559 45.62762
通过membership_prob属性,画图表示个体的稳定性。
# 打印membership_prob
head(cl$membership_prob)
[1] 0.4354753 0.2893287 0.4778663 0.4035933 0.4574012 0.4904582
# 计算群集的数量
num<-length(cl$cluster_scores)
# 从彩虹色中取得对应数量的颜色
rains<-rainbow(num)
cols<-cl$cluster
cols[which(cols==1)]<-rains[1]
cols[which(cols==2)]<-rains[2]
cols[which(cols==3)]<-rains[3]
# 设置透明度,表示个体的稳定性
plot(moons, col=alpha(cols,cl$membership_prob), pch=19)
最后,我们可以在图中,在标记出异常值得分最高的前6个点。
# 对异常值进行排序,取得分最高的
top_outliers <- order(cl$outlier_scores, decreasing = TRUE) %>% head
plot(moons, col=alpha(cols,cl$outlier_scores), pch=19)
text(moons[top_outliers, ], labels = top_outliers, pos=3)
从图中看到,异常得分高的点(outlier_scores)与个体的稳定性(membership_prob),并不是同一类点。异常值通常被认为是,偏离其假定的基础分布的离群点。