卷积网络被大规模应用在分类任务中,输出的结果是整个图像的类标签。然而,在许多视觉任务,尤其是生物医学图像处理领域,目标输出应该包括目标类别的位置,并且每个像素都应该有类标签。另外,在生物医学图像往往缺少训练图片。所以,Ciresan等人训练了一个卷积神经网络,用滑动窗口提供像素的周围区域(patch)作为输入来预测每个像素的类标签。这个网络有两个优点:
第一,输出结果可以定位出目标类别的位置;
第二,由于输入的训练数据是patches,这样就相当于进行了数据增广,解决了生物医学图像数量少的问题。
但是,这个方法也有两个很明显缺点。
第一,它很慢,因为这个网络必须训练每个patch,并且因为patch间的重叠有很多的冗余(冗余会造成什么影响呢?卷积核里面的W,就是提取特征的权重,两个块如果重叠的部分太多,这个权重会被同一些特征训练两次,造成资源的浪费,减慢训练时间和效率,虽然说会有一些冗余,训练集大了,准确率不就高了吗?可是你这个是相同的图片啊,重叠的东西都是相同的,举个例子,我用一张相同的图片训练20次,按照这个意思也是增大了训练集啊,可是会出现什么结果呢,很显然,会导致过拟合,也就是对你这个图片识别很准,别的图片就不一定了)。
第二,定位准确性和获取上下文信息不可兼得。大的patches需要更多的max-pooling层这样减小了定位准确性(为什么?因为你是对以这个像素为中心的点进行分类,如果patch太大,最后经过全连接层的前一层大小肯定是不变的,如果你patch大就需要更多的pooling达到这个大小,而pooling层越多,丢失信息的信息也越多;小的patches只能看到很小的局部信息,包含的背景信息不够。
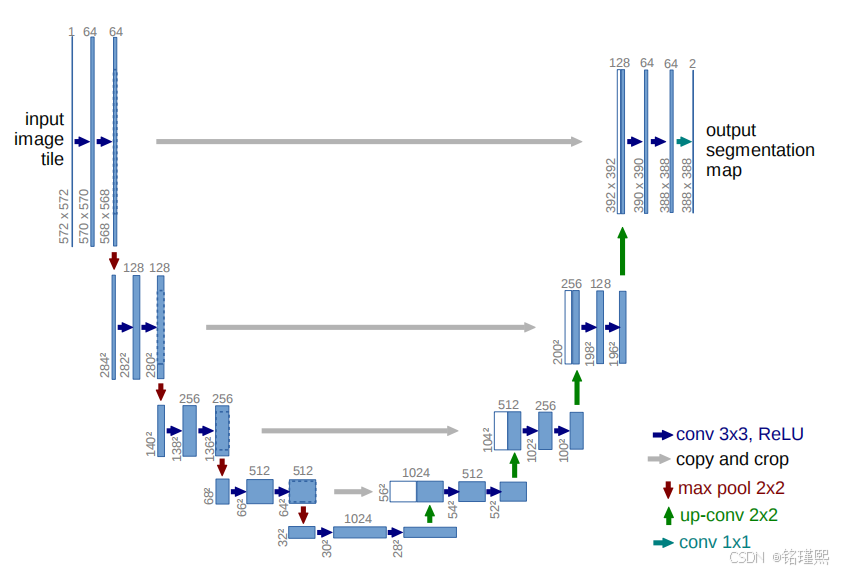
这篇论文建立了一个更好全卷积方法。我们定义和扩展了这个方法它使用更少的训练图片但产生更精确的分割。
(1) 使用全卷积神经网络。(全卷积神经网络就是卷积取代了全连接层,全连接层必须固定图像大小而卷积不用,所以这个策略使得,你可以输入任意尺寸的图片,而且输出也是图片,所以这是一个端到端的网络。)
(2) 左边的网络是收缩路径:使用卷积和maxpooling。
(3) 右边的网络是扩张路径:使用上采样产生的特征图与左侧收缩路径对应层产生的特征图进行concatenate操作。(pooling层会丢失图像信息和降低图像分辨率且是不可逆的操作,对图像分割任务有一些影响,对图像分类任务的影响不大,为什么要做上采样?因为上采样可以补足一些图片的信息,但是信息补充的肯定不完全,所以还需要与左边的分辨率比较高的图片相连接起来(直接复制过来再裁剪到与上采样图片一样大小),这就相当于在高分辨率和更抽象特征当中做一个折衷,因为随着卷积次数增多,提取的特征也更加有效,更加抽象,上采样的图片是经历多次卷积后的图片,肯定是比较高效和抽象的图片,然后把它与左边不怎么抽象但更高分辨率的特征图片进行连接)。
(4) 最后再经过两次反卷积操作,生成特征图,再用两个1X1的卷积做分类得到最后的两张heatmap,例如第一张表示的是第一类的得分,第二张表示第二类的得分heatmap,然后作为softmax函数的输入,算出概率比较大的softmax类,选择它作为输入给交叉熵进行反向传播训练。
下面是U-Net模型的代码实现:(贡献者:黄钦建-华南理工大学)
def get_unet():
inputs = Input((img_rows, img_cols, 1))
conv1 = Conv2D(32, (3, 3), activation='relu', padding='same')(inputs)
conv1 = Conv2D(32, (3, 3), activation='relu', padding='same')(conv1)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
# pool1 = Dropout(0.25)(pool1)
# pool1 = BatchNormalization()(pool1)
conv2 = Conv2D(64, (3, 3), activation='relu', padding='same')(pool1)
conv2 = Conv2D(64, (3, 3), activation='relu', padding='same')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
# pool2 = Dropout(0.5)(pool2)
# pool2 = BatchNormalization()(pool2)
conv3 = Conv2D(128, (3, 3), activation='relu', padding='same')(pool2)
conv3 = Conv2D(128, (3, 3), activation='relu', padding='same')(conv3)
pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)
# pool3 = Dropout(0.5)(pool3)
# pool3 = BatchNormalization()(pool3)
conv4 = Conv2D(256, (3, 3), activation='relu', padding='same')(pool3)
conv4 = Conv2D(256, (3, 3), activation='relu', padding='same')(conv4)
pool4 = MaxPooling2D(pool_size=(2, 2))(conv4)
# pool4 = Dropout(0.5)(pool4)
# pool4 = BatchNormalization()(pool4)
conv5 = Conv2D(512, (3, 3), activation='relu', padding='same')(pool4)
conv5 = Conv2D(512, (3, 3), activation='relu', padding='same')(conv5)
up6 = concatenate([Conv2DTranspose(256, (2, 2), strides=(
2, 2), padding='same')(conv5), conv4], axis=3)
# up6 = Dropout(0.5)(up6)
# up6 = BatchNormalization()(up6)
conv6 = Conv2D(256, (3, 3), activation='relu', padding='same')(up6)
conv6 = Conv2D(256, (3, 3), activation='relu', padding='same')(conv6)
up7 = concatenate([Conv2DTranspose(128, (2, 2), strides=(
2, 2), padding='same')(conv6), conv3], axis=3)
# up7 = Dropout(0.5)(up7)
# up7 = BatchNormalization()(up7)
conv7 = Conv2D(128, (3, 3), activation='relu', padding='same')(up7)
conv7 = Conv2D(128, (3, 3), activation='relu', padding='same')(conv7)
up8 = concatenate([Conv2DTranspose(64, (2, 2), strides=(
2, 2), padding='same')(conv7), conv2], axis=3)
# up8 = Dropout(0.5)(up8)
# up8 = BatchNormalization()(up8)
conv8 = Conv2D(64, (3, 3), activation='relu', padding='same')(up8)
conv8 = Conv2D(64, (3, 3), activation='relu', padding='same')(conv8)
up9 = concatenate([Conv2DTranspose(32, (2, 2), strides=(
2, 2), padding='same')(conv8), conv1], axis=3)
# up9 = Dropout(0.5)(up9)
# up9 = BatchNormalization()(up9)
conv9 = Conv2D(32, (3, 3), activation='relu', padding='same')(up9)
conv9 = Conv2D(32, (3, 3), activation='relu', padding='same')(conv9)
# conv9 = Dropout(0.5)(conv9)
conv10 = Conv2D(1, (1, 1), activation='sigmoid')(conv9)
model = Model(inputs=[inputs], outputs=[conv10])
model.compile(optimizer=Adam(lr=1e-5),
loss=dice_coef_loss, metrics=[dice_coef])
return model