本人是
win11,薛定谔版本是12.9。
官网:https://www.schrodinger.com/
本篇文章的示例大分子蛋白PDB ID为4KNN,小分子配体的MOL ID为MOL004004。
本文部分图源来自知乎https://zhuanlan.zhihu.com/p/416698194,推荐为原作者贡献阅读量捏。
1.Ligand docking讲解(可跳过)
在右上角的操作区的tasks搜索Ligand docking,打开面板如下:
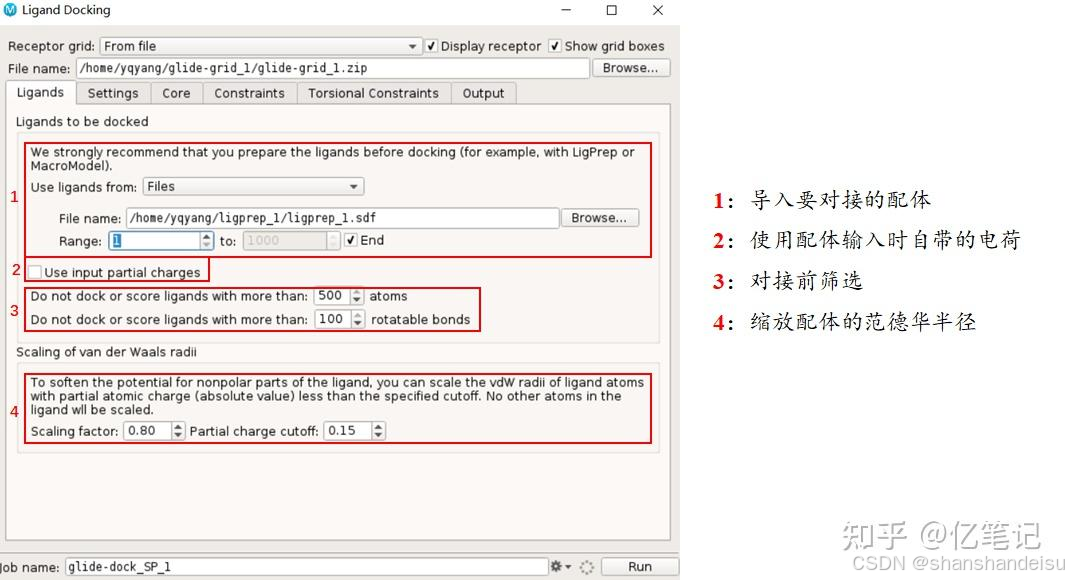
1.1.Ligands
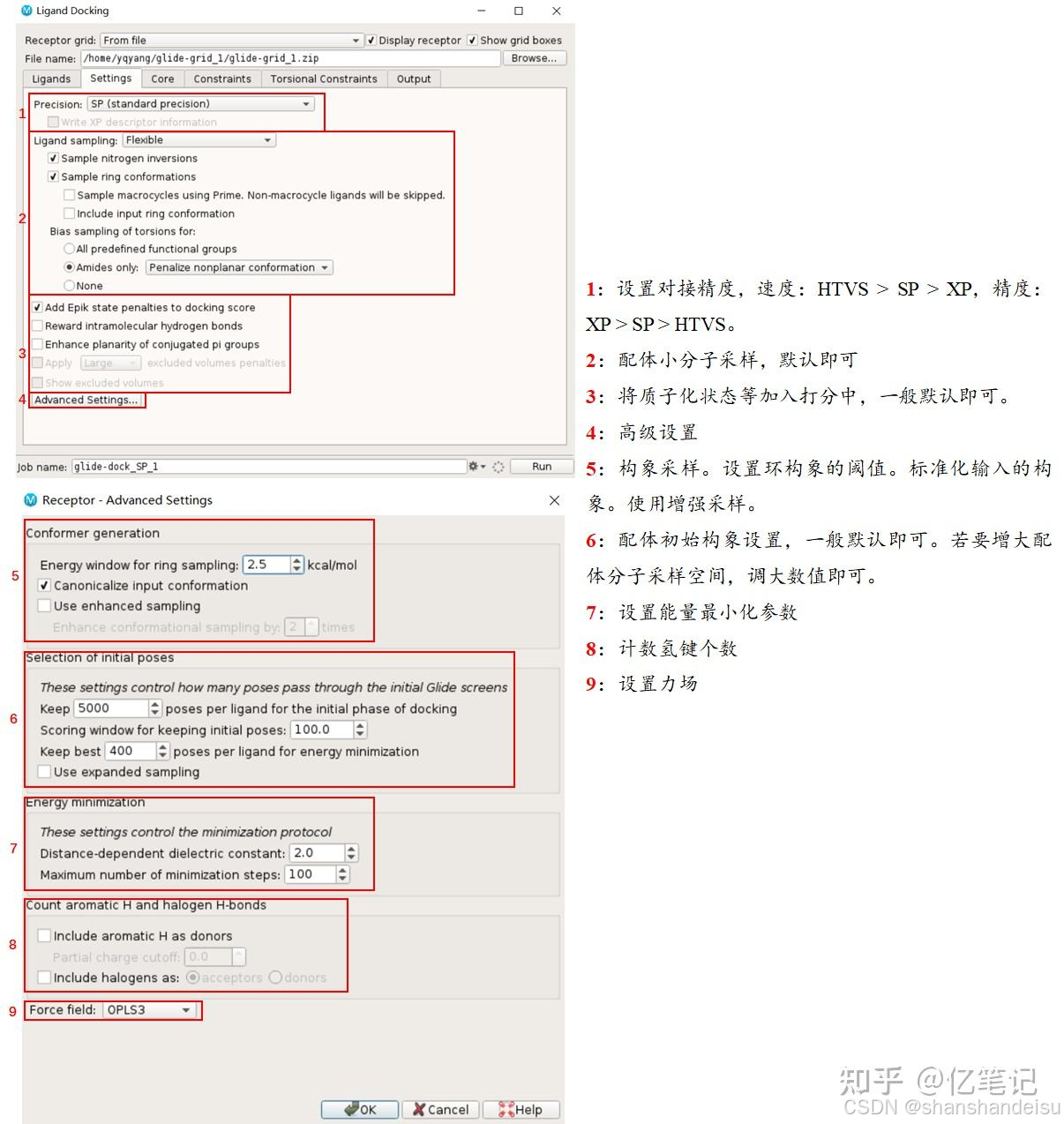
1.2.Settings
HTVS:高通量筛选,一般用来筛选很多个小分子SP:和HTVS算法原理差不多,但是会降低采样的彻底性。XP:开始和SP是一样的,但是采样更严格,运行时间比SP长。对于形状互补更严格,所以一般用于排除假阳性。但是需要额外的license
补充:如果你需要虚拟筛选一个很大的数据库,建议先用
SP,按照打分排序,取前10%-30%,用XP重新对接。并勾选write xp descriptor information。
还有一个virtual screening模块,相当于先用HTVS、再sp、再xp。
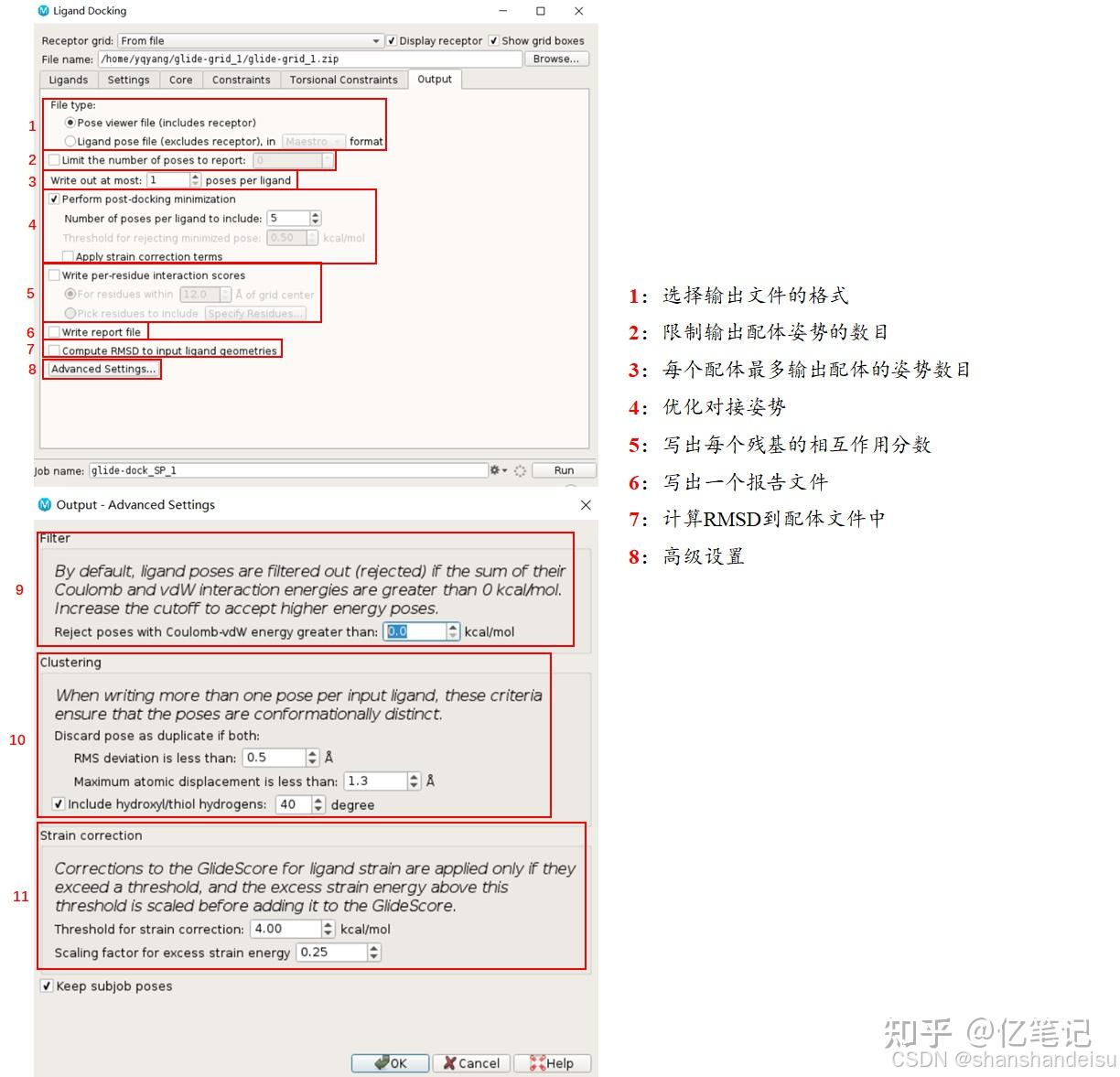
1.3.Output
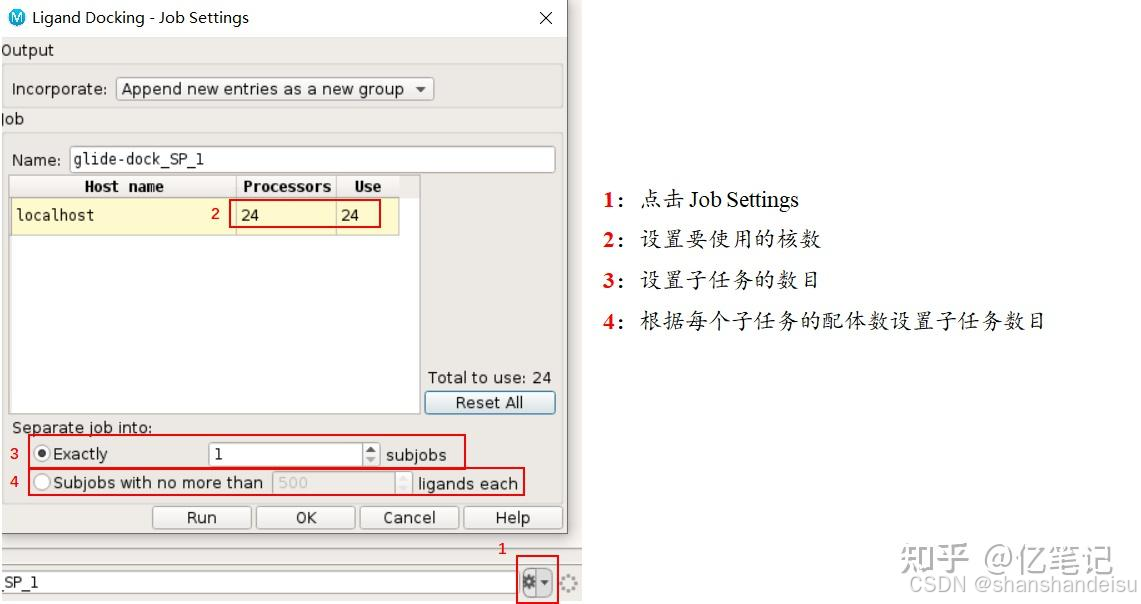
1.4.job settings
2.Ligand docking实战
在右上角的操作区的tasks搜索Ligand docking,打开面板如下:
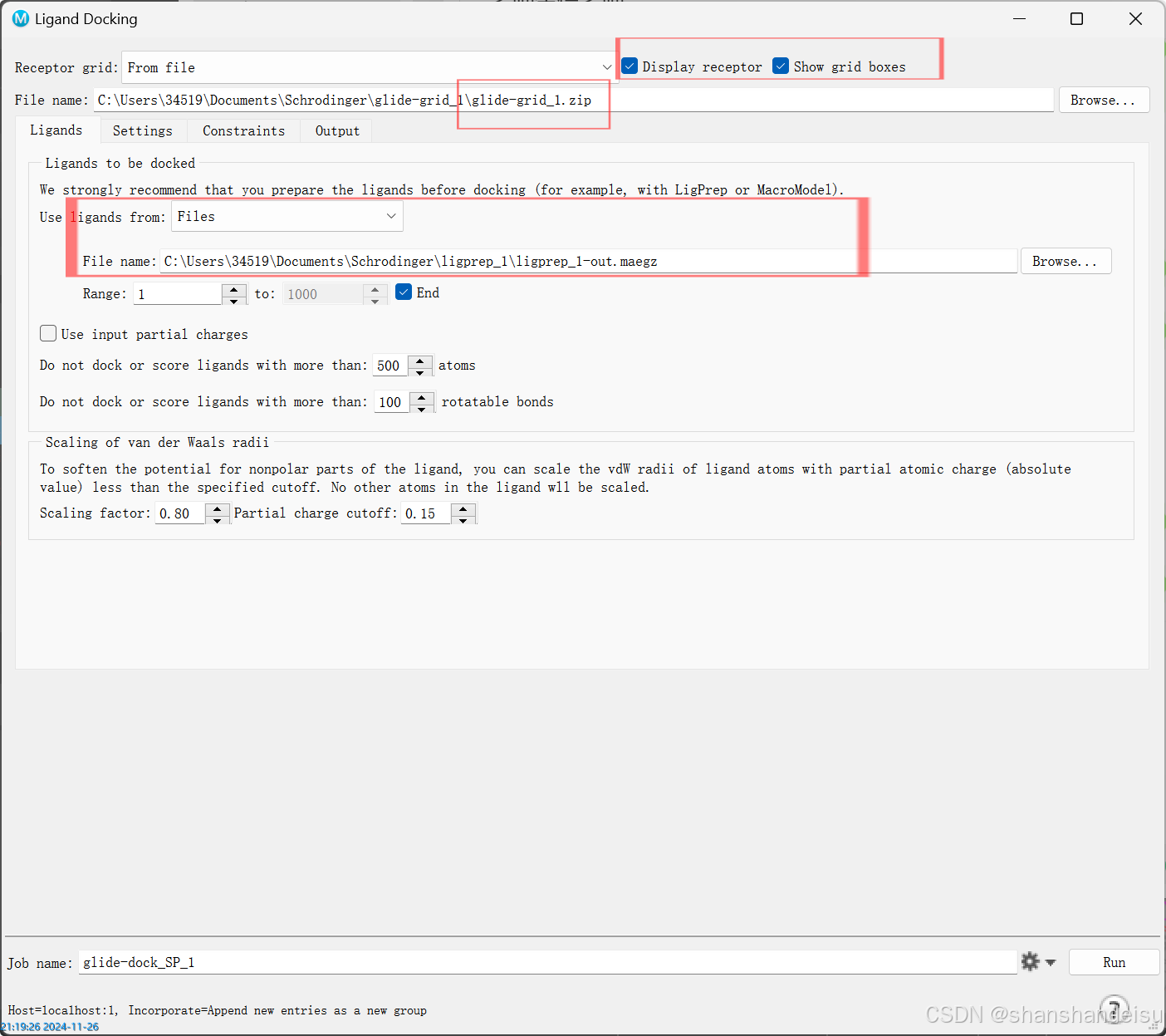
默认从文件中加载glide-grid1.zip并勾选display receptor 和show grid boxes。
选择配体:use ligands from files,并加载ligprep_1-out.maegz。最后直接run即可。
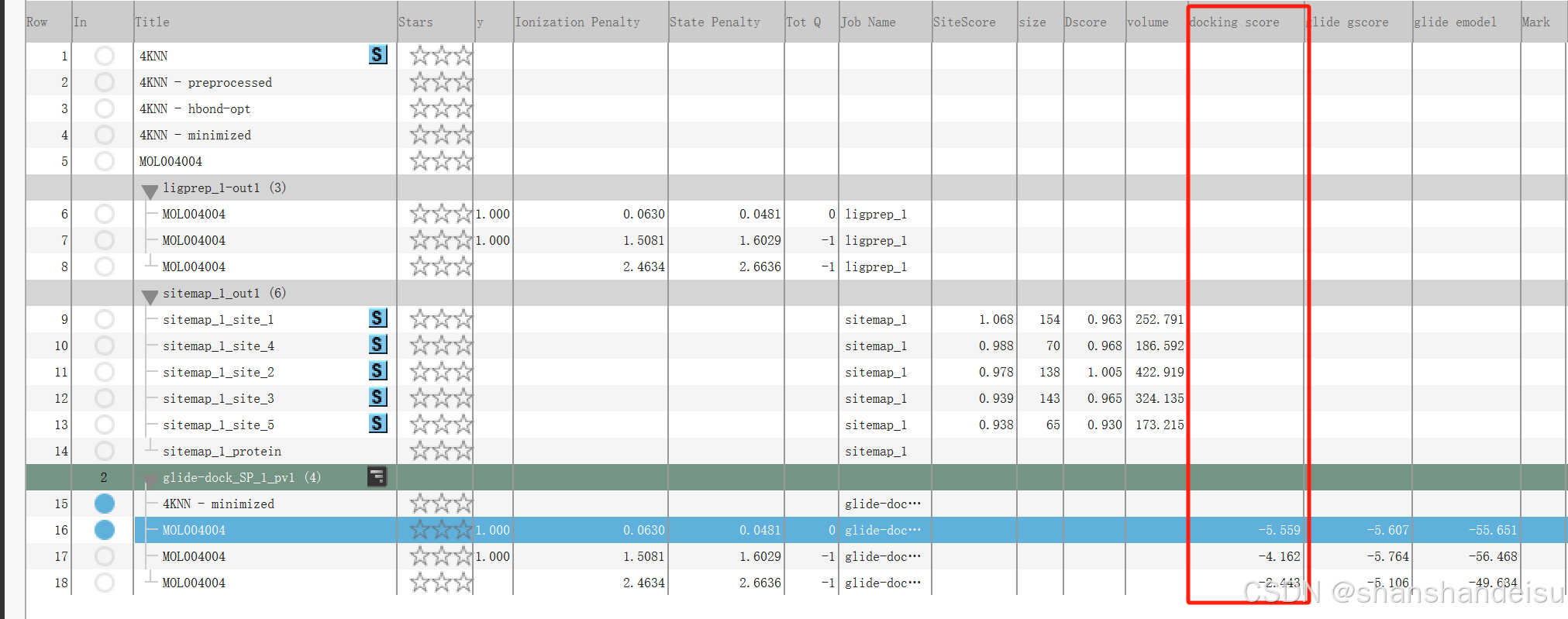
对接完成后会得到glide-dock_SP_1_pv.maegz文件,且左侧工作区会生成多个名字和小分子配体名字一样的条目,实际上对接后的构象,在我这是MOL004004。
我们点击操作区的table,显示如下,只用看docking score这一列。
可以看到,最好的结果是-5.559,只能说有作用力,但对接得不算特别好。
3.可视化
待更ing