Nature | 用于科学药物发现的分子视频衍生基础模型
药物发现是一个复杂且耗时的过程,涉及药物靶点识别、分子设计、合成及药效验证。近年来,随着分子表征学习的发展,基于机器学习和深度学习的方法为药物发现提供了新思路。然而,如何准确地捕获分子的三维结构及其动态变化仍然十分困难。本文提出了一种创新的分子视频基础模型——VideoMol,通过将分子的动态构象转化为视频数据,结合自监督学习策略,实现分子动态特征与理化信息的高效表征。在43个药物发现基准数据集上的实验结果显示,VideoMol 在靶点结合活性预测、分子性质预测、抗 SARS-CoV-2 活性识别以及虚拟筛选任务中均表现出显著的性能优势。
创新点与优势
VideoMol 的核心创新在于引入了分子视频表征。与传统的分子表征(如分子指纹、分子图结构和静态图像)不同,VideoMol 将分子的三维构象动态变化表示为视频,通过旋转分子三维结构生成 60 帧视频,捕获不同视角下的结构信息。这种动态视频表示方法有效地解决了静态方法中因视角遮挡和信息丢失导致的局限性。与传统的分子图神经网络(GNN)方法相比,VideoMol因能够提取更丰富的三维信息因而在学习分子动态特征方面更具优势。此外,VideoMol 提出了三种自监督学习任务,包括视频感知预训练(VAP)、方向感知预训练(DAP) 和 化学感知预训练(CAP)。通过 VAP 任务,模型可以最大化同一分子视频帧之间的相似性,并最小化不同分子视频帧之间的相似性,从而提高模型的鲁棒性。DAP 任务通过学习分子帧之间的旋转关系,使模型能够推断三维结构的方向变化,增强空间理解能力。CAP 任务则通过分子的化学子结构和理化特征进行聚类,进一步强化模型的化学信息捕获能力。这三种任务的结合使 VideoMol 能够从多个角度全面地学习分子的动态和化学信息,显著提升分子表征的准确性。
在任务适用性方面,VideoMol 展现了出色的泛化能力,能够在多种药物发现任务中取得优异表现。具体来说,VideoMol 在药物靶点结合活性预测任务中,针对激酶和 GPCR 蛋白等靶点,AUC 和 RMSE 等指标均显著优于现有的基准方法。在分子性质预测方面,VideoMol 在溶解度、脂溶性、毒性等指标上均取得了领先性能。此外,在抗 SARS-CoV-2 活性预测任务中,VideoMol 相较于传统方法表现出更高的准确性和稳定性。在虚拟筛选与分子对接任务中,VideoMol 成功筛选出具有较高结合亲和力的抑制剂,在靶向 BACE1、COX-1 和 COX-2 等靶点的药物的虚拟筛选中,其性能远超传统的分子对接方法,充分证明了 VideoMol 在分子三维动态信息捕获方面的优势。
实验结果与性能表现
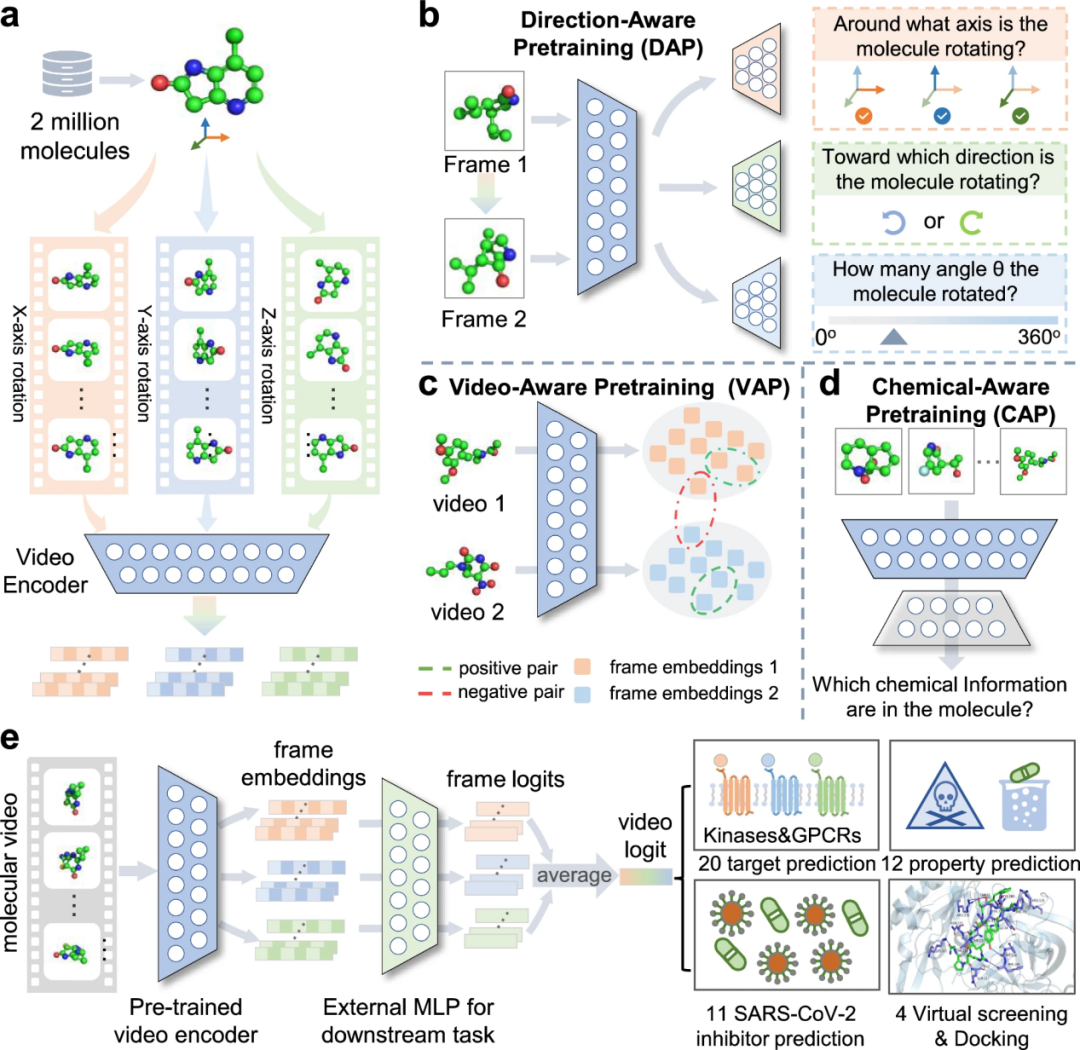
图1:VideoMol 基础模型概述
a 分子视频的特征提取。首先,作者在 3D 空间结构中渲染了 200 万个带有构象异构体的分子。然后,作者围绕x,y,z轴并为 Molecule 视频的每一帧生成快照。最后,作者将分子框架馈送到视频编码器中以提取潜在特征。b–d 用于预训练视频编码器的三个自我监督任务。方向感知预训练 (DAP) 任务用于通过使用轴分类器(橙色)、旋转分类器(绿色)和角度分类器(蓝色)来区分分子框架对之间的关系(如旋转轴、旋转方向和旋转角度)。视频感知预训练 (VAP) 任务用于最大化视频内相似度和最小化视频间相似度。化学感知预训练 (CAP) 任务用于通过使用化学分类器(灰色)来识别分子视频中与物理化学结构相关的信息。e VideoMol 在下游基准(如结合活性预测和分子特性预测)上的微调。在预训练视频编码器之后添加多层感知器 (MLP),用于微调四种类型的下游药物发现任务(20 个靶点预测、12 个特性预测、11 个 SARS-CoV-2 抑制剂预测和 4 个虚拟筛选和对接)。作者将每帧的结果 (logits) 组装起来,作为分子视频 (video logit) 的预测结果。
通过在多个基准数据集上的实验,VideoMol 的性能得到了全面验证。在靶点结合活性预测任务中,VideoMol 在 10 个激酶结合活性预测任务上的 AUC 提升了 5.9%,部分数据集提升高达 20.3%。在 GPCR 结合活性预测任务中,VideoMol 在 RMSE 和 MAE 指标上分别降低了 4.5%-6.2%。在分子性质预测任务中,VideoMol 在溶解度、脂溶性、毒性等指标上显著优于传统方法。在抗 SARS-CoV-2 活性预测任务中,VideoMol 的平均性能较现有方法提升了 3.9%-8.1%。此外,在虚拟筛选任务中,VideoMol 对 BACE1 等靶点的识别准确率远超传统分子对接方法,进一步证明了其在药物筛选任务中的应用潜力。
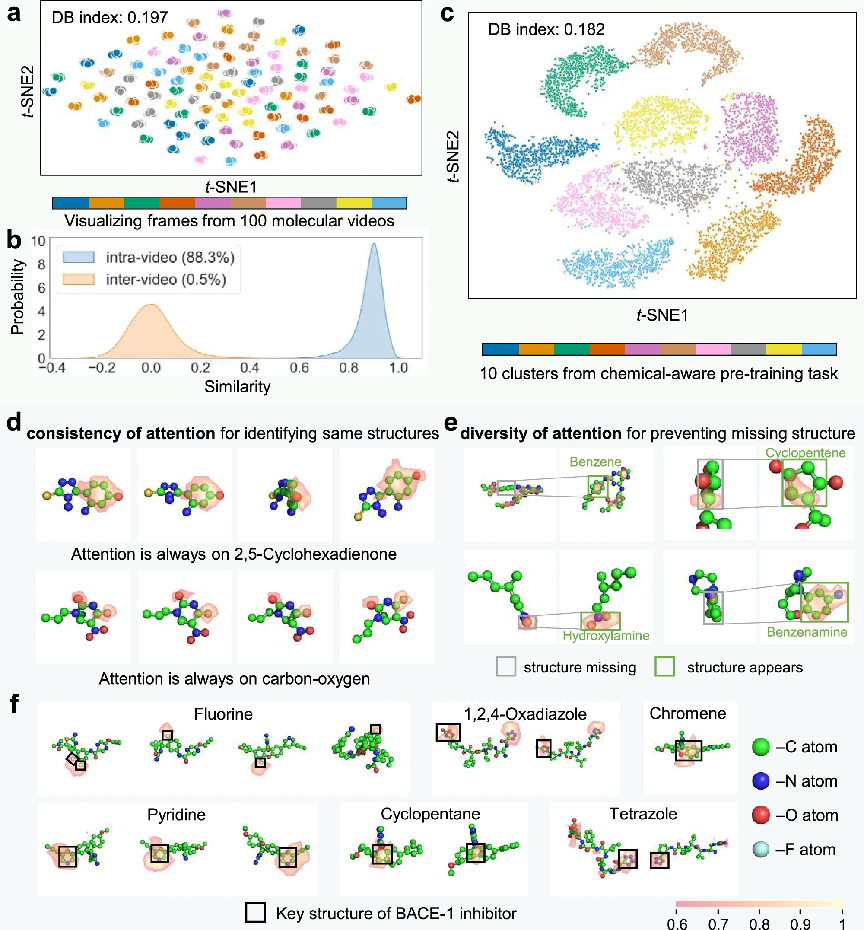
图2:VideoMol 的生物学解释和特征分布
a 100 个分子视频中每帧的可视化(每个视频 60 帧)。表示由 VideoMol 提取,并通过 t-SNE 进行尺寸缩小。不同的颜色表示不同群集视频中的帧。DB index 是评估集群质量的指标,该值越大,集群性能越好。b 视频内和视频间的相似性分布(n = 20,000 个样本)。相似度是使用视频内或视频间的一对帧计算的。括号中的内容表示分布的平均相似性。通过 VideoMol 提取的特征的 c t-SNE 可视化(10,000 个样本)。不同的颜色代表不同的聚类标签(这个聚类标签是在化学感知预训练任务中获得的)。VideoMol 在分子框架上的 d-f Grad-CAM 可视化。我们用 0.6 作为可视化的阈值,即将低于 0.6 的重要性设置为 0。在 d 中,每行代表一个分子视频。在 e 中,分子框架对分别表示结构缺失的框架和结构出现的框架。在 f 中,每个面板代表来自不同分子框架的与 BACE-1 抑制活性相关的关键结构的示例。
结论与展望
VideoMol 通过引入分子视频表示和三种自监督学习任务,为分子表征学习提供了一种全新的解决方案。相比于现有的分子表征方法,VideoMol 能够更加全面地捕获分子的动态特征和理化信息,在药物发现任务中表现出卓越的性能和广泛的适用性。未来研究可以进一步优化模型计算效率,结合分子动力学信息,探索更高效的分子表征方法。此外,VideoMol 的框架具有极高的扩展性,可与其他分子表征方法(如序列、图结构)相结合,构建更为全面的分子表征模型。
综上所述,VideoMol 在分子表征学习和药物发现领域具有革命性创新,突破了传统静态表示的局限,为基于人工智能的药物发现提供了新的思路和强大的工具。
参考资料:Xiang, H., Zeng, L., Hou, L. et al. A molecular video-derived foundation model for scientific drug discovery. Nat Commun 15, 9696 (2024). https://doi.org/10.1038/s41467-024-53742-z