本文将对R语言中自带的heart数据集进行分析。本文会包含所有代码,尽可能多的使用有关可视化的函数以及数据分析的模型。
一、研究概述
1.1 数据集简介
数据集来源:使用R语言 glmpalh 包中自带的数据heart.data
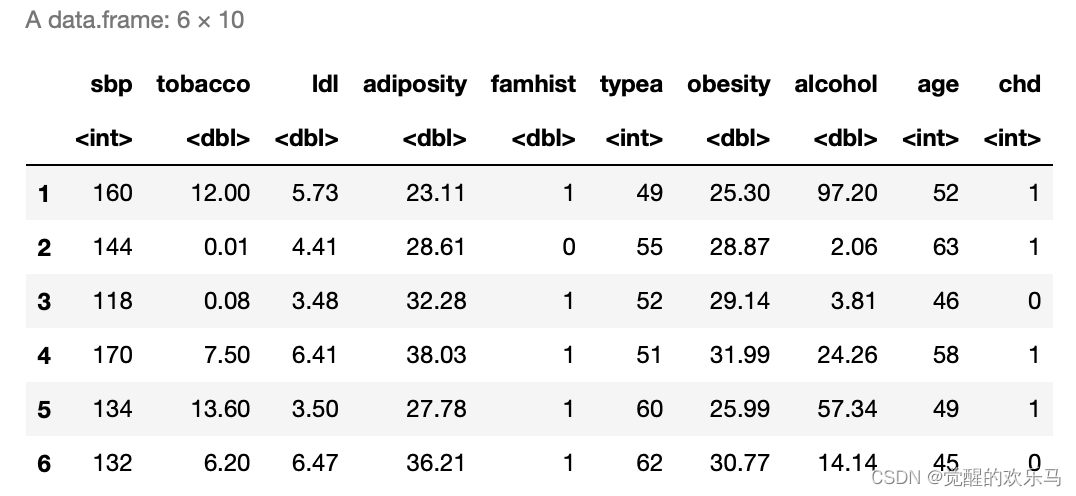
数据集内容:该数据集描述了452个南非人的身体健康状况指标,用来研究哪些因素对是否患有心脏病有影响。其中,因变量chd是一个个二分类变量,代表是否患有冠心病,自变量共9个,包括 sbp(血压)、tobacco(累计烟草量)、ldl(低密度脂蛋白胆固醇)、 adiposity(肥胖)、famhist(是否有心脏病家族史,定性变量)、typea(型表现)、obesity (过度肥胖)、alcohol (当前饮酒)、age(年龄)。
1.2 研究内容
我们可以调查这些因素,探究那些变量会对是否患有冠心病有影响,并对于这些影响因素对人提出适当建议。通过给定个人数据来预测此人是否患有冠心病(仅做参考)。
二、实例操作
2.1 数据预处理
2.1.1 导入数据
首先导入ncvreg包,取出heart.data数据集,命名为data,并简单查看数据。
#导入数据
library(ncvreg)
data(heart)
data = heart
head(data)2.1.2数据清洗
我们首先对data进行探索,寻找空值,以免对后文产生影响,并将类别型变量转换为因子格式
#查看数据集概述
dim(data)
#查看空值数量
table(is.na(data))
#转换格式
data$famhist = as.factor(data$famhist)
data$chd = as.factor(data$chd)我们可以发现数据集一共有462行数据,没有空值
2.1.3划分训练集和测试集
这里我们选择80%的数据为训练集,命名为data_tr;20%为测试集,命名为data_te
同时设置一个种子,模型每次数据一样
#划分数据集
set.seed(6)
index = sample(nrow(data),nrow(data)*0.8)
data_tr = data[index,]
data_te = data[-index,]2.2构建模型
2.2.1Logistic回归
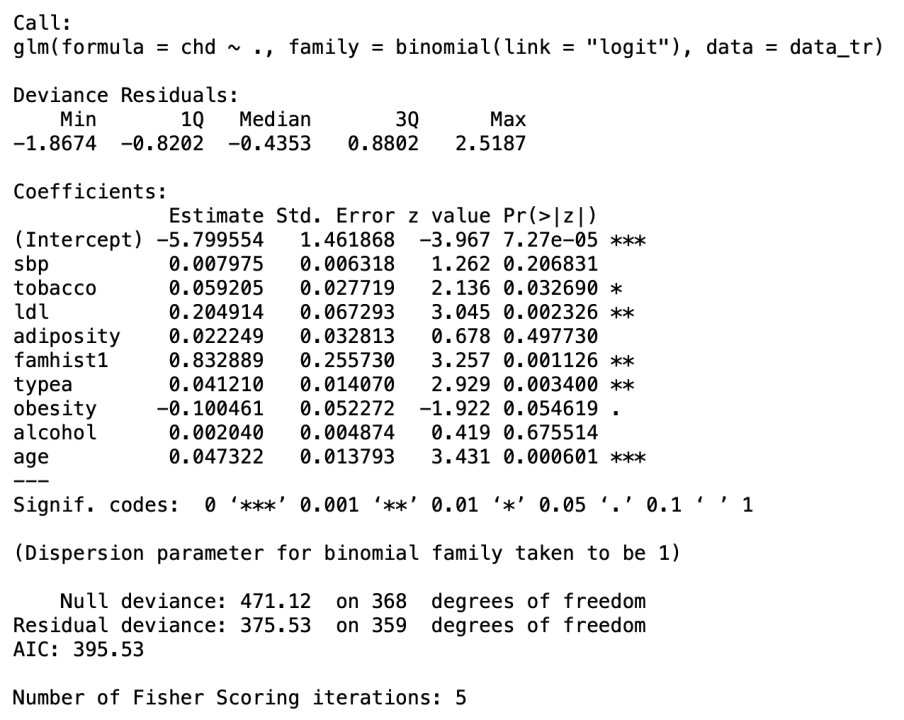
我们通过glm()函数构建模型,数据采用训练集,让机器选择最优的参数。
#Logistic回归
set.seed(6)
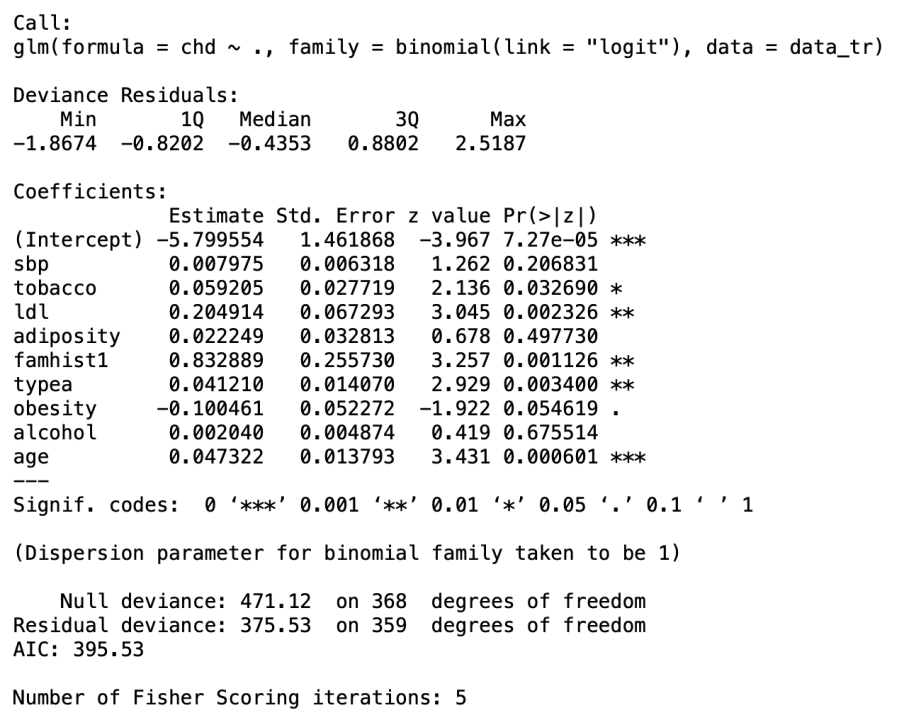
data.log = glm(chd~.,family=binomial(link="logit"),data = data_tr)
#预测
data.log.pred = predict(data.log, data_te, type="response")
data.log.class = rep(0,nrow(data_te))
data.log.class[data.log.pred>0.5]=1 #以0.5作为阈值
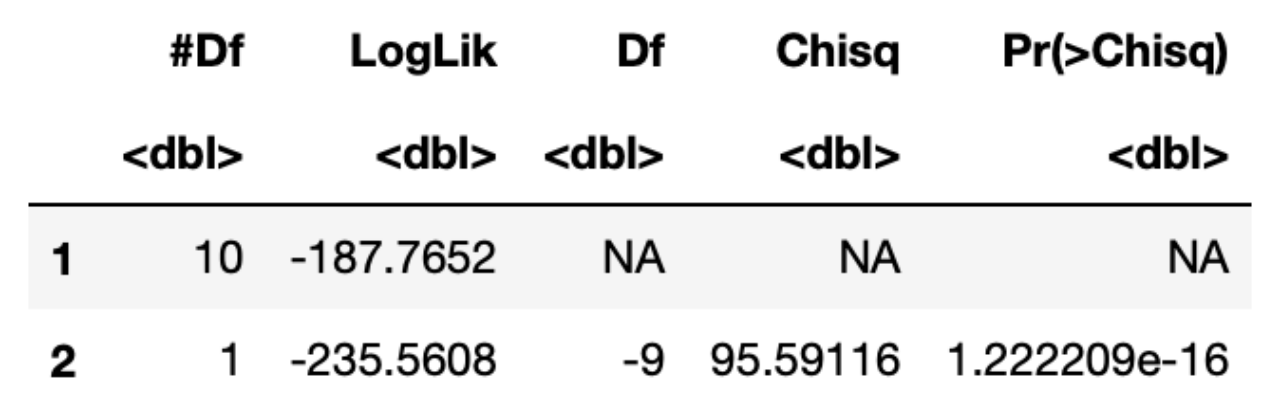
table(data.log.class,data_te$chd)随后我们通过LR检验(LR=2(lnL-lnL0) 似然比检验)来验证模型是否显著。
L对应第一行LogLik的值,为-187.76;L0为第二行LogLik的值,为-234.56;LR值为Chisq,为96.59,它对应的p值只有1.2e-16,因此模型整体是显著的,有参考意义。
最后我们通过混淆矩阵计算此模型的准确率,通过由训练集计算出的模型,对测试集进行预测,通过自变量的值来预测此人是否患有冠心病。
这里输出的混淆矩阵,我们可以计算出此模型的准确率是(49+19)/(49+17+8+19)。最后结果为73.12%
2.2.2决策树
首先我通过tree()函数构建决策树
#决策树
library(tree)
data.tree = tree(chd~.,data_tr)
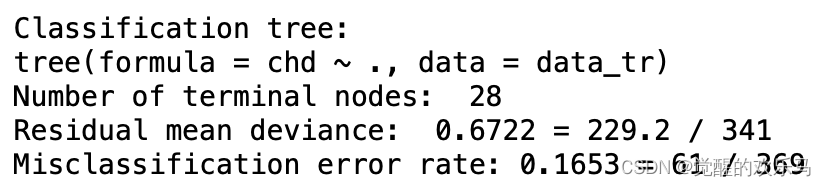
summary(data.tree)
我们可以使用summary()函数显示生成的节点数和准确率。目前决策树的错误率是16.53%
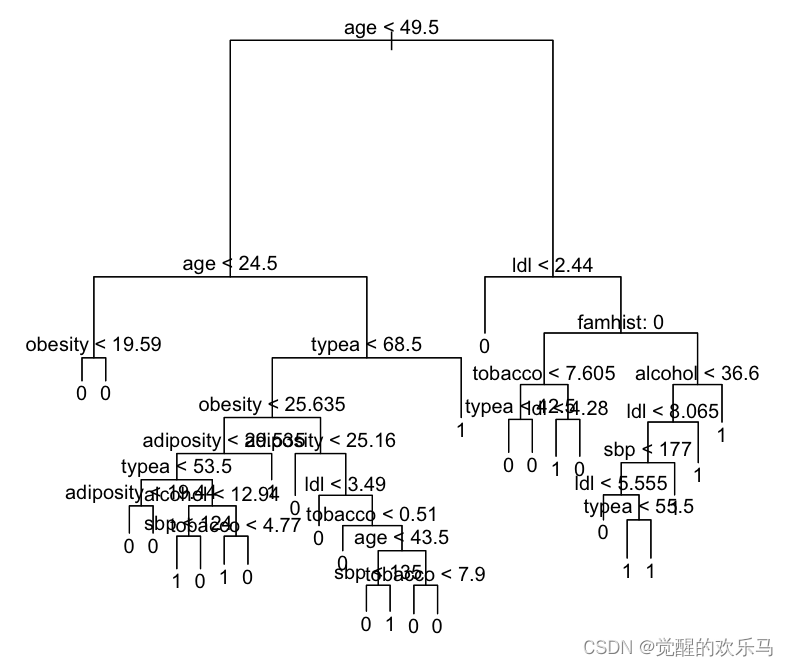
我们可以通过函数plot画出树的结构。text函数显示节点结构,其中参数pretty=0可以输出定性自变量的类别名。
#未剪枝的树
plot(data.tree)
text(data.tree,pretty=0)随后通过predict函数评估次数的预测效果。在分类树的情况下,参数type="class"令R语言返回真实的预测类别
#预测
tree.pred = predict(data.tree,data_te,type = "class")
table(tree.pred,data_te$chd)这里输出的混淆矩阵,我们可以计算出此模型的准确率是(44+11)/(44+25+13+11)。最后结果为59.14%
接下来,考虑剪枝能否改进预测结果,用函数cv.tree0进行交叉验证以确定最优的树的复杂性
#计算修剪
set.seed(6)
cv.data.tree = cv.tree(data.tree,FUN = prune.misclass)
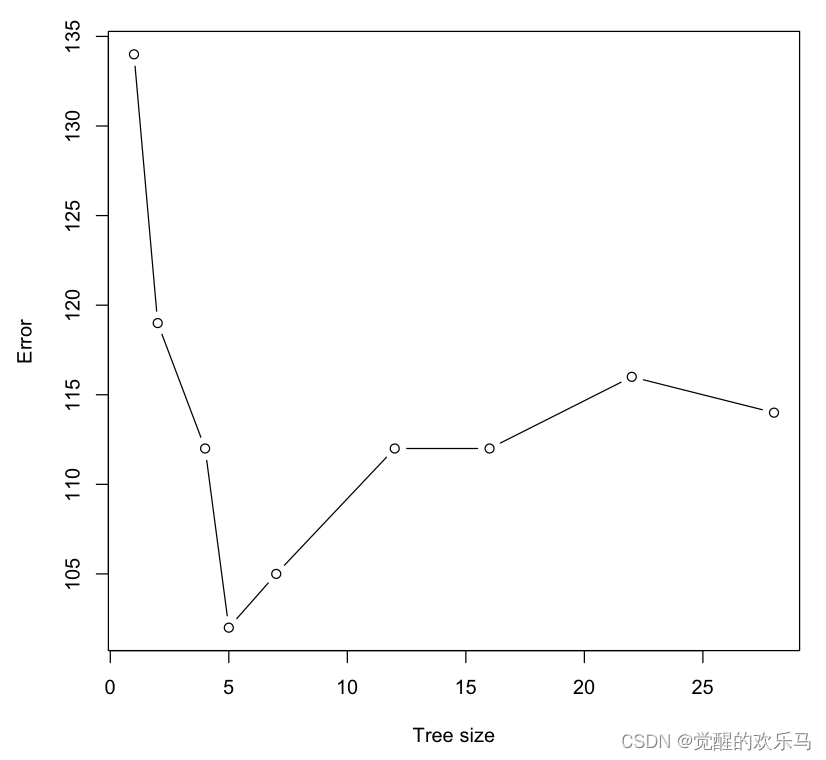
cv.data.treedev反映的是交叉验证错误率,size表示终端节点数。当节点数为5时,交叉验证错误率最低
通过plot函数查看选择节点
plot(cv.data.tree$size,cv.data.tree$dev,type="b",xlab="Tree size",ylab="Error")
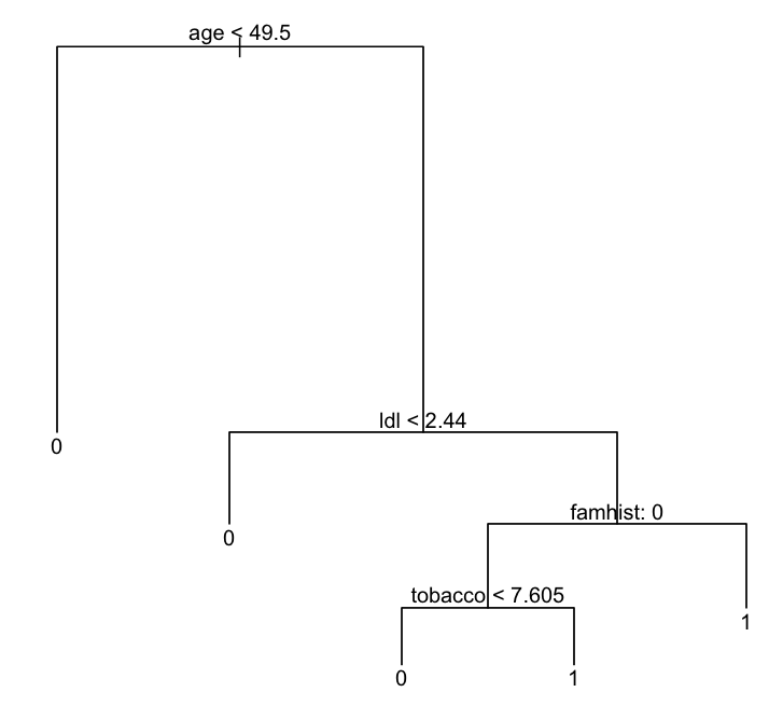
#终端节点数为5时交叉验证错误最低且树更加简洁这里选择5个节点数可以使树更简洁,通过prune.misclass函数进行剪枝。随后再次计算准确率
#剪枝
prune.data = prune.misclass(data.tree,best=5)
plot(prune.data)
text(prune.data,pretty=0)
#再次计算准确率
tree.cv.pred = predict(prune.data,data_te,type = "class")
table(tree.cv.pred,data_te$chd)这里输出的混淆矩阵,我们可以计算出此模型的准确率是(52+15)/(52+15+21+5)。最后结果为72.04%
在通过剪枝操作后,可以发现树的结构变简单了,准确率也大幅度提升了。
2.2.3随机森林
#随机森林
library(randomForest)
set.seed(6)
data.rf = randomForest(chd~.,data = data_tr , impportance = TRUE)
#预测
data.rf.pred = predict(data.rf,newdata = data_te,type="class")
table(data.rf.pred,data_te$chd)
我们可以发现,在通过随机森林构造模型后,准确率同样为72.04%
2.2.4模型评估 ROC曲线和AUC值
在构建完三个模型后,我们进一步通过ROC曲线和AUC值寻找最优的模型,另外两个模型成为次模型。
#3.3.4 ROC曲线比较模型的好坏,计算模型的AUC值
library(ROCR)
library(pROC)
rocplot = function(pred, truth, ...) {
predob = prediction (pred, truth)
perf = performance (predob, "tpr", "fpr")
plot(perf, ... )
auc = performance(predob,"auc")
auc = unlist(slot(auc,"y.values"))
auc = round(auc, 4)#保留4位小数
text(x = 0.8,y = 0.1, labels = paste("AUC =", auc))
}
#输出结果变为概率值
set.seed(1)
data.log.pred2 = predict(data.log, data_te, type="response")
data.lda.pred2 = predict(data.lda,data_te) $ posterior[,2]
data.qda.pred2 = predict(data.qda,data_te) $ posterior[,2]
cv_roc = roc(data_te$chd,as.numeric(tree.cv.pred))
rf_roc = roc(data_te$chd,as.numeric(data.rf.pred))
#输出ROC曲线和AUC值
par(mfrow = c(2,3))
y = data_te$chd
rocplot(data.log.pred2,y,main="Logit")
plot(cv_roc, print.auc=TRUE, auc.polygon=TRUE, max.auc.polygon=TRUE, print.thres=TRUE,main='tree')
plot(rf_roc, print.auc=TRUE, auc.polygon=TRUE, max.auc.polygon=TRUE, print.thres=TRUE,main='forest')
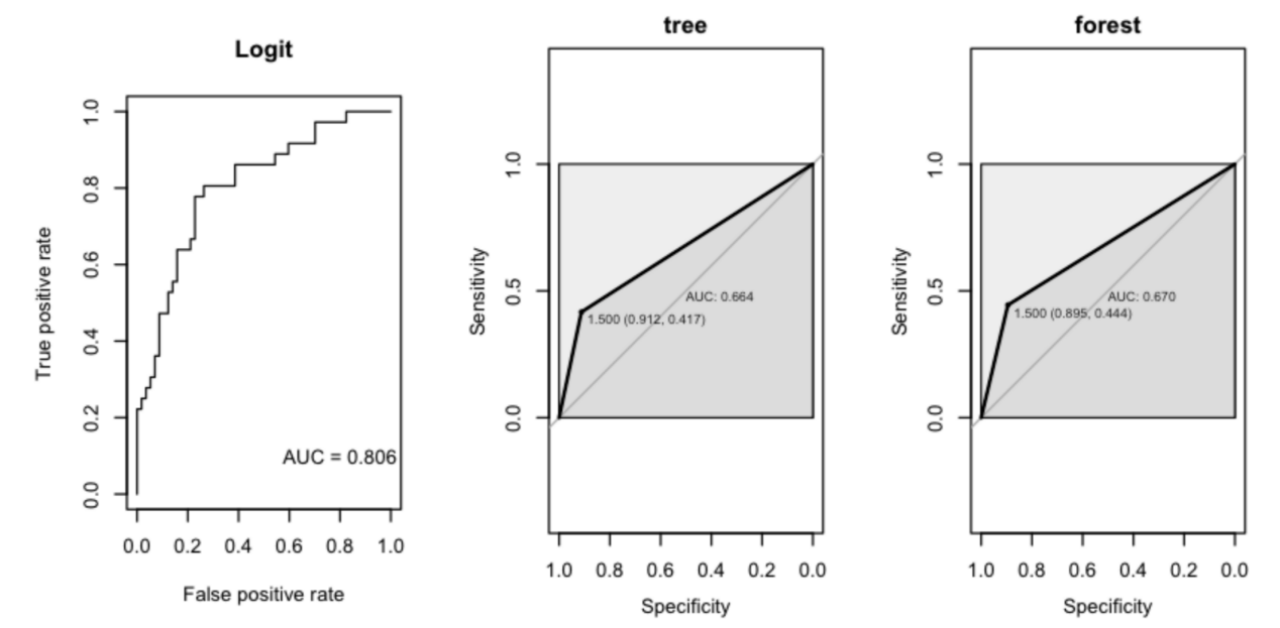
这里我们可以发现Logistic的模型AUC值最高,为0.806;决策树和随机森林的AUC值分别为0.664和0.670。因此最后我们以Logsitic模型为主模型进行分析。
三、结论
首先观察Logistic模型的结果。
Estimate:回归系数(b0,以上显示为-5.8),即与每个预测变量相关的β系数估计(如sbp的系数为0.08)
Std.Error:系数估计值的标准误。这代表了系数的准确性。标准误差越大,估计值的准确性越低。
z value:z统计量,即系数估计值(第2列Estimate)除以估算值的标准误(第3列Std.Error)
Pr(>|z|):对应于z统计量的p值。p值越小,估计值对结果变量来说越重要。
可以看出,在9个预测变量中,只有5个与结果显着相关。这些包括:tobacco(累计烟草量)、ldl(低密度脂蛋白胆固醇)、 famhist(是否有心脏病家族史,定性变量)、typea(型表现)、age(年龄)。
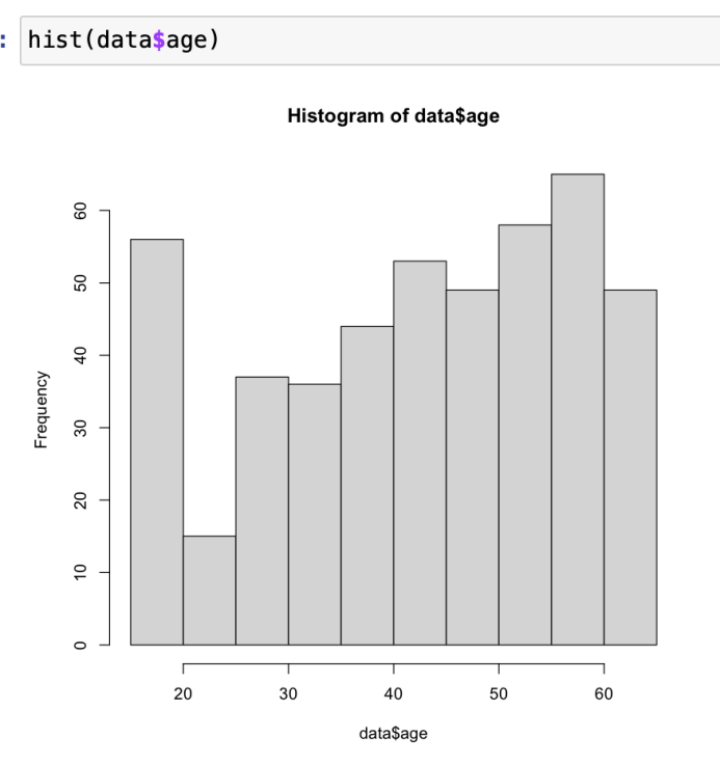
随后我们继续对如上变量进行数据可视化操作。我们首先画出了年龄的直方图,可以发现数据中20-25年龄段的人较少,因此对于这部分人群的预测可能准确率不如其他年龄段。
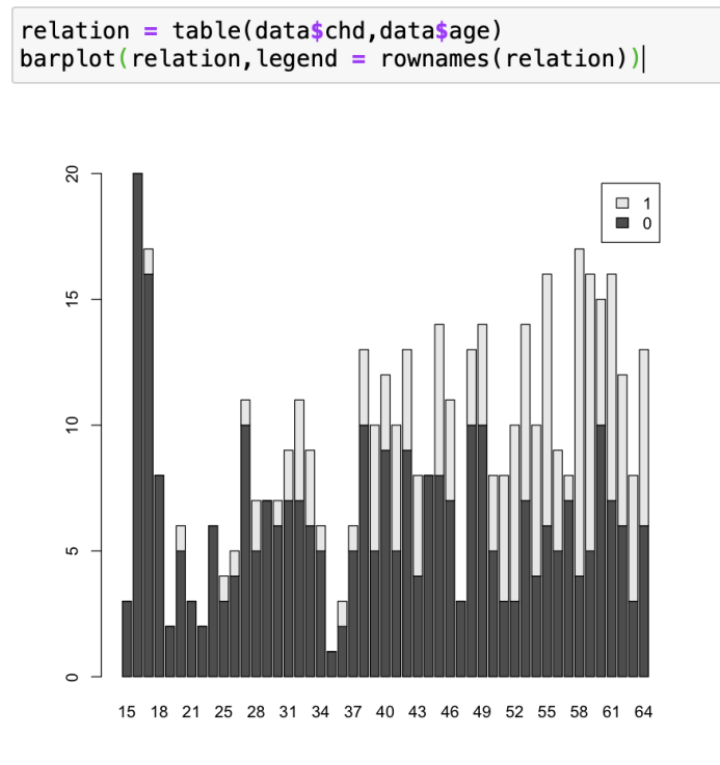
随后我们通过条形图绘制年龄和是否患有冠心病的关系
这里很明显的可以发现,在随着年龄提升的同时,患有冠心病的人也越来越多,符合我们上述模型所得出的结论。
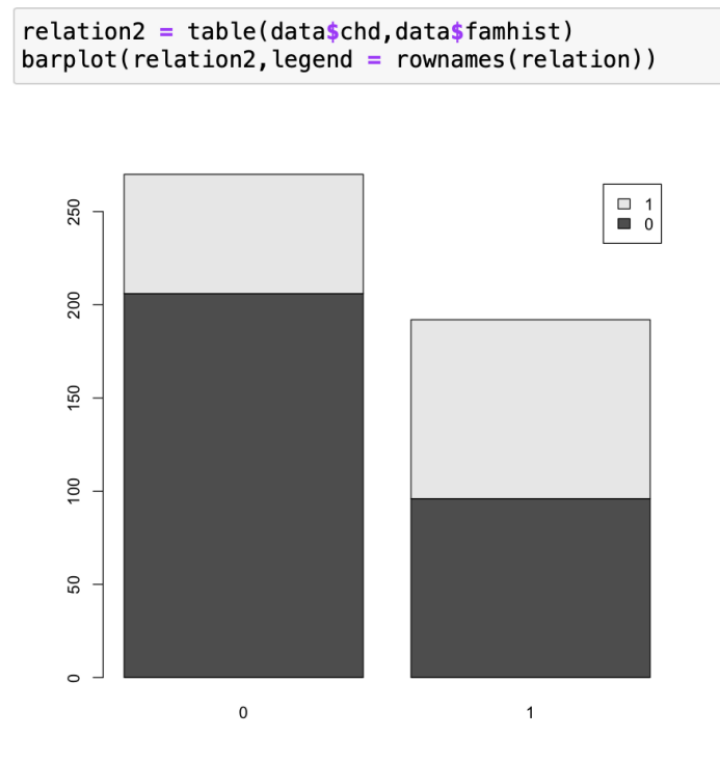
我们继续对下一个有关变量进行可视化。调查famhist(是否有心脏病家族史)这一定性变量的影响。通过图像,我们可以发现当famhist为0,也就是没有心脏病家族史的时候,患有冠心病的概率大约为30%,而famhist为1时,也就是有心脏病家族史的时候,患有冠心病的概率大约50%。因此对于是否有心脏病家族史对于是否会患有冠心病也有很大影响
此外的剩余变量这里就不做详细说明了。
在这里我们就可以实现我们研究目的的第一个要点了,给人们提供建议。人们需要着重关注于以下几个点:tobacco(累计烟草量)、ldl(低密度脂蛋白胆固醇)、 famhist(是否有心脏病家族史,定性变量)、typea(型表现)、age(年龄)。减少累计烟草量能够减少患病的概率,对于有心脏病家族史和年龄大的人可以去医院进行检查,提前发现症状提早治疗。
此外,模型的另一个功能即为预测,也可以称为机器诊断。我们可以将病人的数据输入,通过模型给出结论,是否患有冠心病。
这里我们随机给个例子,根据此人的数据我们可以发现机器预测此人没有冠心病。但由于准确率只有大约70%,并且不同结果阈值的设置同样导致准确率变动的问题。所以预测结果有待考证。我们可以通过在边预测边收集数据的方式逐步提升模型的准确率,或者构建不同计算过程的模型,当两个或多个模型均给出同样的答案后,才能提升真正的预测准确率。