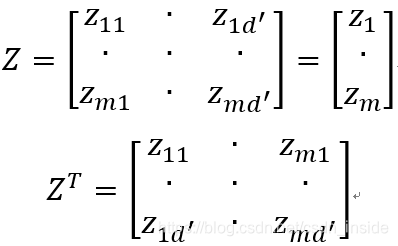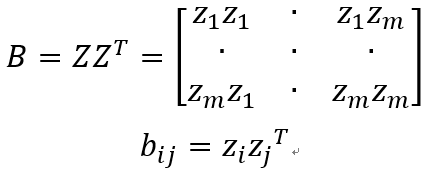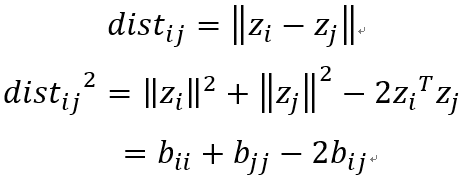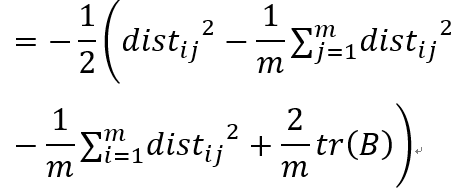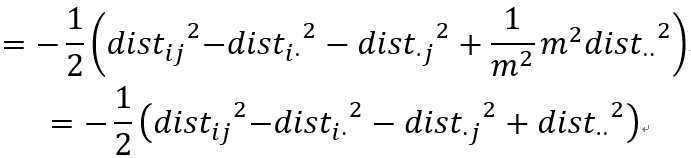MDS的思路就是保持新空间与原空间的相对位置关系,先用原空间的距离矩阵D,求得新空间的内积矩阵B,再由内积矩阵B求得新空间的表示方法Z。
西瓜书上的推导比较简洁,自己详细的推了一遍。
假设Z矩阵是新空间的表示,mxd'维,z1,z2到zm表示一个行向量,即每一个样本。
定义新空间的内积矩阵B,B矩阵的bij元素,即是z的第i行与第j行的内积。
为了使新空间与原空间保持相对位置关系,新空间两个样本的距离要等于(或者约等于)原空间的距离。
为了便于讨论,这里使Z被中心化,即Z所有元素相加=0。
下面开始推导,对于里面的bij项,对i从1到m求和,j不动:
同理:
对于bii项,对的i从1到m求和,注意这里是对i求和,不对j求和,所以结果是mbjj:
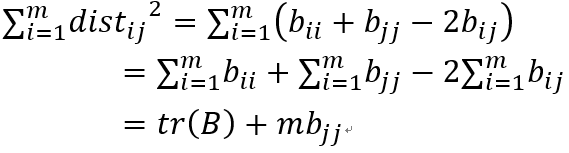
tr(B)表示B矩阵的迹,对角元素之和。
同理,对j求和:
················②
再用上式对i求和:
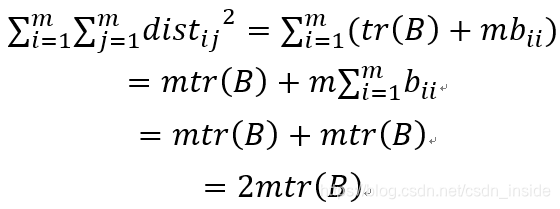
为了化简,令
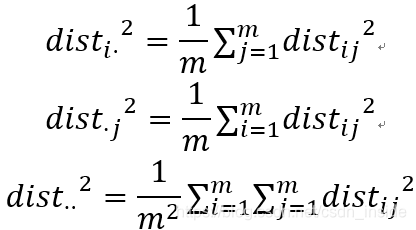
由①②式,移项可得:
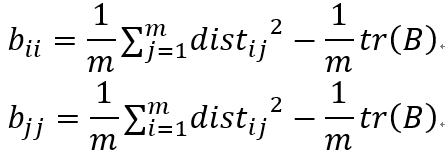
由③式,移项可得:
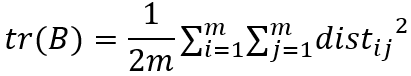
由
可得:
将⑦⑧式的bii和bjj带入上式,可得:
再由④⑤式带入,替换掉、
,⑨带入替换掉tr(B),可得:
最后由⑥化简最后一项得到:
这样就可以由原空间距离矩阵D求得内积矩阵B。
由于
其中Λ是特征值组成的对角阵,就可得到:
即用B求得Z。
代码+实战
import numpy as np
import matplotlib.pyplot as plt
class MyMDS:
def __init__(self,n_components):
self.n_components=n_components
def fit(self,data):
m,n=data.shape
dist=np.zeros((m,m))
disti=np.zeros(m)
distj=np.zeros(m)
B=np.zeros((m,m))
for i in range(m):
dist[i]=np.sum(np.square(data[i]-data),axis=1).reshape(1,m)
for i in range(m):
disti[i]=np.mean(dist[i,:])
distj[i]=np.mean(dist[:,i])
distij=np.mean(dist)
for i in range(m):
for j in range(m):
B[i,j] = -0.5*(dist[i,j] - disti[i] - distj[j] + distij)
lamda,V=np.linalg.eigh(B)
index=np.argsort(-lamda)[:self.n_components]
diag_lamda=np.sqrt(np.diag(-np.sort(-lamda)[:self.n_components]))
V_selected=V[:,index]
Z=V_selected.dot(diag_lamda)
return Z
先用自己的MDS,然后用sklearn的MDS,可视化一下iris.data:
from sklearn.datasets import load_iris
iris=load_iris()
clf1=MyMDS(2)
iris_t1=clf1.fit(iris.data)
plt.scatter(iris_t1[:,0],iris_t1[:,1],c=iris.target)
plt.title('Using My MDS')
from sklearn.manifold import MDS
clf2=MDS(2)
clf2.fit(iris.data)
iris_t2=clf2.fit_transform(iris.data)
plt.scatter(iris_t2[:,0],iris_t2[:,1],c=iris.target)
plt.title('Using sklearn MDS')降维的效果基本一致,对于紫色的花,都可以有效区分;对于绿色和黄色的花,有一些难以区分。
再对sklearn的digits数据降维可视化看一看:
from sklearn.datasets import load_digits
digits=load_digits()
clf3=MyMDS(2)
digits_t1=clf3.fit(digits.data)
plt.scatter(digits_t1[:,0],digits_t1[:,1],c=digits.target,
alpha=.5)
plt.colorbar()
plt.title('Using My MDS')
from sklearn.manifold import MDS
mds=MDS(n_components=2)
mds.fit(digits.data)
digits_t2=mds.fit_transform(digits.data)
plt.scatter(digits_t2[:,0],digits_t2[:,1],c=digits.target,
alpha=.5)
plt.colorbar()
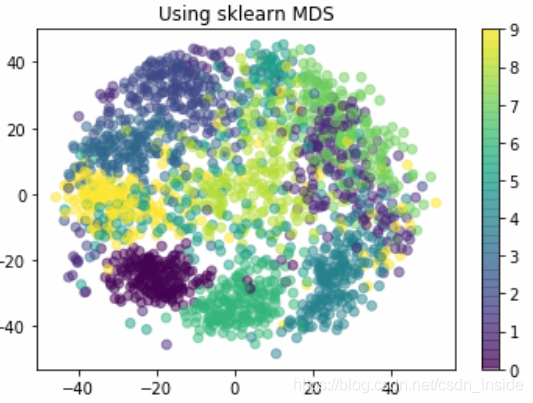
plt.title('Using sklearn MDS')
from sklearn.decomposition import PCA
pca=PCA(n_components=2)
pca.fit(digits.data)
digits_t2=pca.fit_transform(digits.data)
plt.scatter(digits_t2[:,0],digits_t2[:,1],c=digits.target
,alpha=.5)
plt.colorbar()
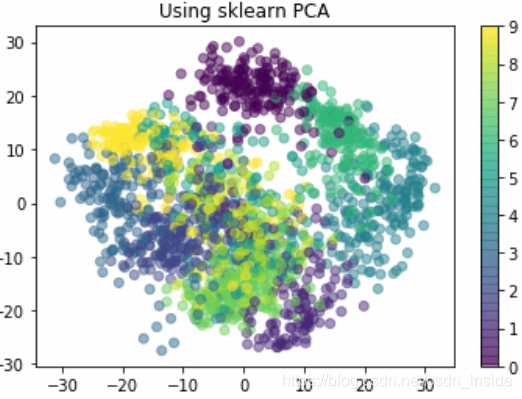
plt.title('Using sklearn PCA')
from sklearn.manifold import Isomap
iso=Isomap(n_components=2)
iso.fit(digits.data)
digits_projected=iso.transform(digits.data)
plt.scatter(digits_projected[:,0],digits_projected[:,1],c=digits.target
,alpha=.5)
plt.colorbar()
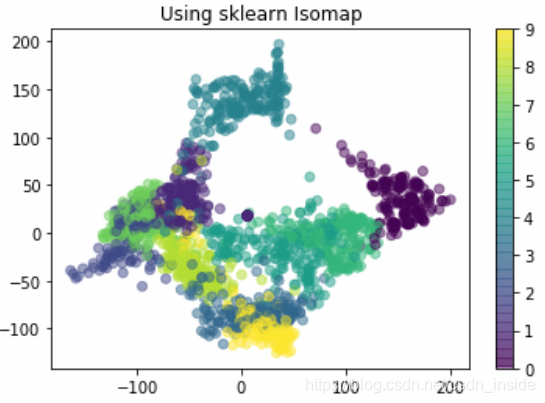
plt.title('Using sklearn Isomap')sklearn的MDS跑了很久,不知道为何。。。
效果。。。三者小巫见大巫,都不是很好,有的数字难以区分。
所以换Isomap流形学习看看:
发现效果好比上述两种降维要一些,说明digits数据集的真实分布,不是线性的,更接近于流形,单纯的PCA和MDS不能很好的解决问题。