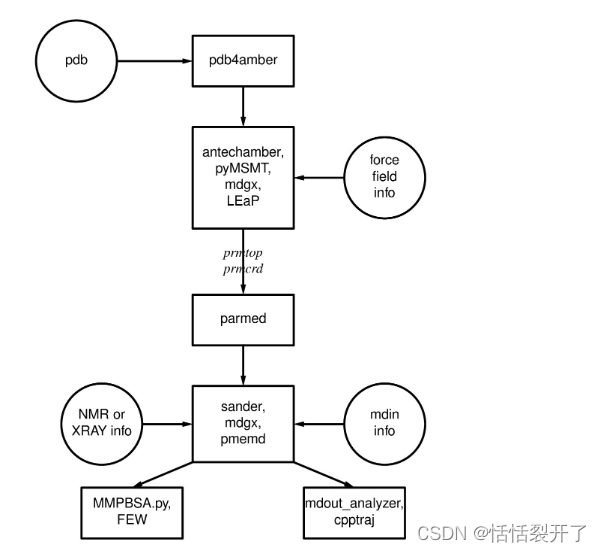
总体介绍
各类模块作用:
pdb4amber:通常可以帮助准备来自其他地方(如rcsb.org)的pdb格式的文件,以与LEaP兼容。
LEaP:是在Amber中创建新系统或修改现有系统的主要程序。它可以作为命令行程序tleap或GUI xleap使用。它结合了早期版本的Amber的准备、链接、编辑和parm的功能。
Antechamber:是开发小有机分子(如药物,修饰氨基酸)的主要程序,使用一般amber力场(GAFF)版本的力场。这些可以直接在LEaP中使用,也可以作为进一步参数开发的起点。
parmed:提供了一种提取关于在参数拓扑文件中定义的参数的信息的简单方法。它还可以用于检查参数拓扑文件对复杂系统是否有效(请参阅检查有效性命令),而且它还可以对该文件进行简单的修改。
MCPB.py:提供了一种建立、原型和验证金属蛋白和有机金属化合物的MM模型的方法。它使用结合加静电模型来扩展现有的成对相加力场。它是Python中MCPB的重新实现,具有更高效的工作流和以前版本的许多建模过程。
IPMach.py提供了一个工具来促进离子的非键合模型(12-6 LJ模型和12-6-4LJ型模型)的参数化。
一、pdb4amber
pab4amber 1a0h.pdb > 1a0h.amber.pdb
删除氢原子:
pdb4amber -i WT.pdb -o WT_noH.pdb -y --dry
添加氢原子:
reduce WT_noH.pdb > WT_H.pdb
二、LEaP
开始:
tleap
加入力场:
source leaprc.protein.ff14SB
source leaprc.DNA.OL15
source leaprc.lipid17
source leaprc.water.tip3p
source leaprc.gaff2
处理好的文件做一份拷贝:文件名为name
cp name.pdb name.process.pdb
用八面体水盒子溶剂化分子:文件名为name
source leaprc.water.tip3p
name = loadpdb "name.mbr.pdb"
slovateOct name SPCBOX 14.0
中和电荷:文件名为name
charge name
加入电荷,并保存拓扑力场文件以及坐标文件:number/name自行修改
addIons name Cl- (number)
addIons name Na+ (number)
saveamberparm name svt.name.top svt.name.crd
三、建立多肽序列
项目实例:
1.加载适当的力场
tleap
source leaprc.protein.ff14SBonlysc
2.为隐式溶剂 GB 计算指定半径。
set default PBradii mbondi3
3.利用 LEaP 中的序列命令构建奇尼奥林,一种 10 个氨基酸的合成肽。
chig = sequence {GLY TYR ASP PRO GLU THR GLY THR TRP GLY}
4.在 tleap 中保存 pdb 文件。
savepdb chig chignolin_ext.pdb
5.保存一个 prmtop 和 inpcrd 文件 tleap
使用amber运行任何内容都需要拓扑文件和一组起始坐标(最小化、分子动力学等)。此步骤将使用适当的力场参数(parm7)和起始坐标(inpcrd 文件)保存您的单位(chig)。
saveamberparm chig chig_ext.parm7 chig_ext.crd
6.退出
quit
四、在显式溶剂中构建蛋白质系统
实例:
以2yx8.pdb为例
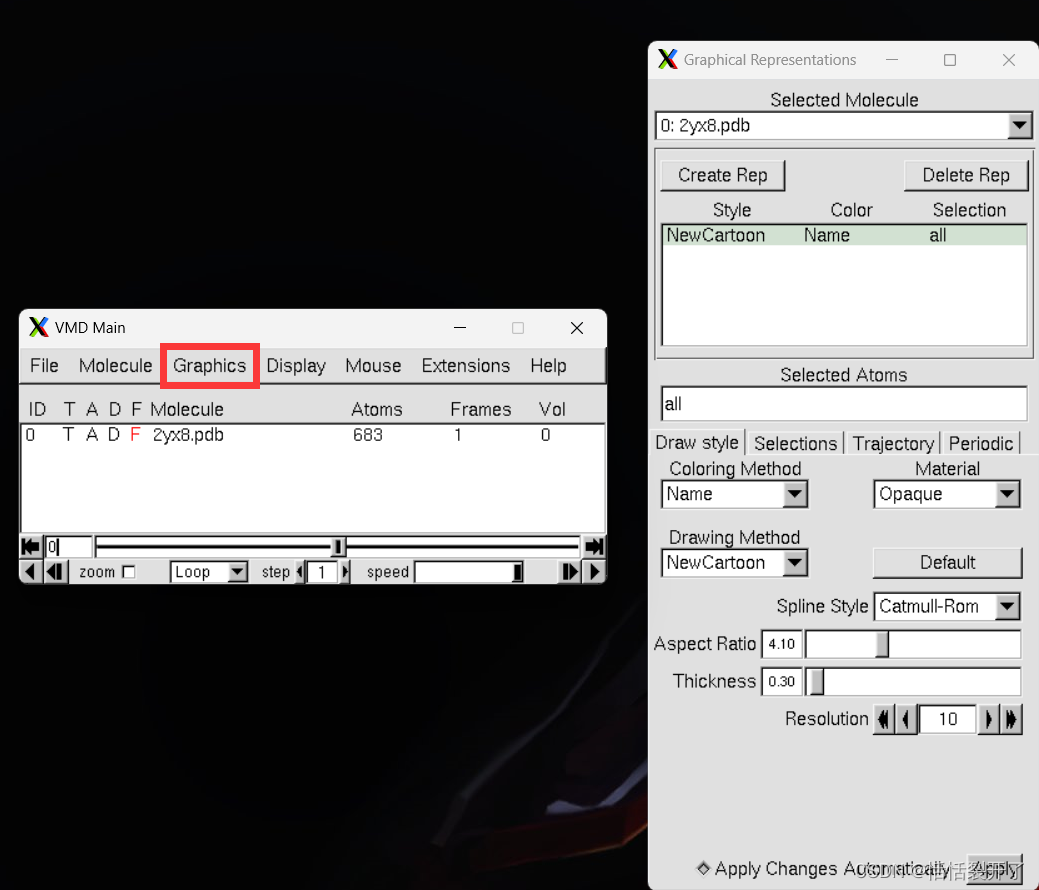
1.使用 VMD 检查蛋白质结构。
VMD下载及相关操作:
VMD的下载安装及基础
VMD与amber的结合应用
vmd 2yx8.pdb
VMD控制面板,显示设置
2.评估和分析要导入amber的蛋白质结构。
2.1复制文件进行后续操作
cp 2yx8.pdb 2yx8_fxMET.pdb
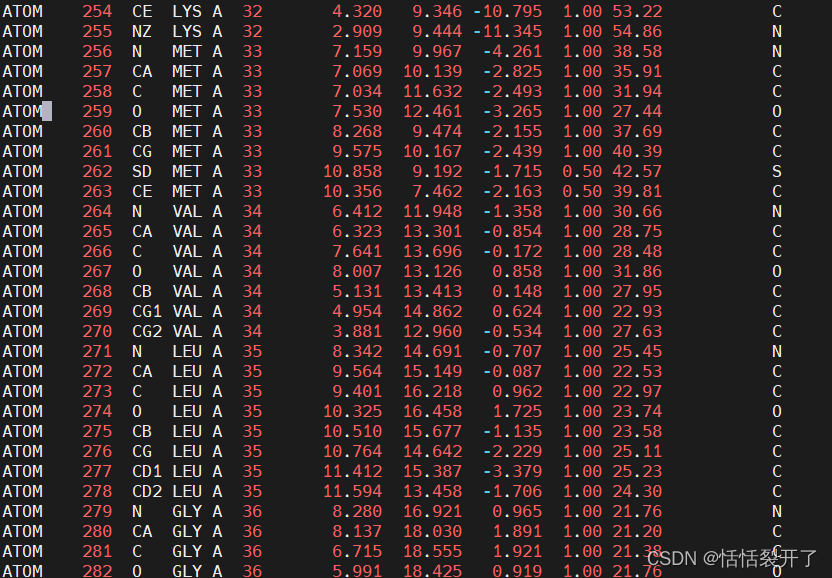
2.2 vi打开修改及编辑,无基础可以参照vi基础教程:vi基础操作教程
vi 1fsc.pdb
打开后界面↓
2.3编辑:
第一列中的HETATM标签意味着这些残基不会与周围的氨基酸相连。 查找/替换HETATM为“ATOM”,以便序列是连续的。确保在 ATOM 之后保留两个空格,以便其余列将排成一排。
第二列是原子数。不要编辑这个!
第三列是原子的名称。例如,CA是α碳。因为蛋氨酸不含硒,我们需要改变这个原子到硫。将原子名称从 SE 编辑为 SD(蛋氨酸中硫原子的原子名称)。将最后一列中的 SE 更改为一个S 也是。
第四列是上面提到的重命名。将所有 MSE 条目更改为 MET。
替换命令:
:s/search/replace/g
缺失电子密度(氨基酸)
您可以在 PDB 文件本身中查找缺失的残留物。例如,如果您缺少残基 15-18,您将看到信息 在 Resid 14 上,然后是 Resid 19。
完成复制蛋白附件
cp 2yx8_fxMET.pdb 2yx8_fxMET_fxCYS.pdb
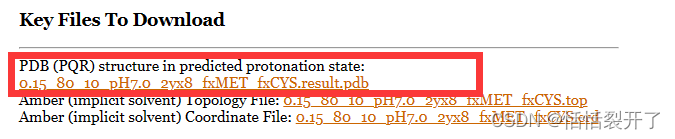
2.4 检查质子化状态
如果给定PDB,程序H++可以预测组氨酸的质子化状态
把PH改成人体PH7.0,点下方process,继续运算
输出结果:
完成
2.5创建 PDB 的_noCONECT副本并删除 CONECT 记录
cp 2yx8_fxMET_fxCYS_fxHIS.pdb 2yx8_fxMET_fxCYS_fxHIS_noCONECT.pdb
2.6以简短的名称保存
cp 2yx8_fxMET_fxCYS_fxHIS_noCONECT.pdb RAMP1.pdb
3.使用 LEaP 在明确的溶剂中构建蛋白质系统。
3.1启动tleap
tleap
3.2荷载力场
source leaprc.protein.ff19SB
3.3将要用的pdb加载到ramp(相当于一个变量)
ramp=loadpdb RAMP1.pdb
保存
saveamberparm ramp RAMP1_ion.prmtop RAMP1_ion.inpcrd
3.4添加电荷
addIons ramp Na+ 2
addIonsRand ramp Na+ 19 Cl- 19